Magnetresonanz-Bildgebung bei zerebralen Gliomen
Universitätsinstitut für Diagnostische und Interventionelle Neuroradiologie<br>Inselspital, Universität Bern<br>E-Mail: arsany.hakim@insel.ch
Die neuroradiologische Bildgebung von Gliomen hat in den letzten Jahren viele bemerkenswerte Veränderungen erfahren. Die neuroradiologische Diagnostik spielt in der Diagnoseunterstützung, der Therapieplanung und im Monitoring des Krankheitsverlaufs unter Radio- und Chemotherapie eine bedeutende Rolle und wird durch Verfahren der automatisierten Bildanalyse zunehmend augmentiert.
Keypoints
-
Neue MR-basierte Bildgebungsmarker unterstützen die Korrelation zwischen bildgebendem Phänotyp und molekularen Markern (2-HG-MRS).
-
Methoden des «advanced imaging» (Perfusion, Diffusion, MRS) erlauben die Abgrenzung zwischen therapieassoziierten Veränderungen und Tumorprogression.
-
Methoden der automatisierten Bildanalyse, wie z.B. die Tumorvolumetrie, unterstützen die longitudinale Verlaufsbeurteilung des Tumorwachstums.
-
Die Entwicklung radiomischer Bildanalysen und molekularer MR-basierter Bildgebungsmarker (APT-CEST) befindet sich derzeit noch in klinischer Erprobung.
Die visuelle Beurteilung struktureller Bilddatensätze wird durch Anwendung funktioneller und metabolischer Verfahren zur Feststellung einer Tumorprogression und zur nicht invasiven Korrelation mit dem zugrunde liegenden Genotyp ergänzt. Diese Entwicklungen werden durch die neuropathologische Klassifikation der Gliome von 2016 unterstützt, in der die Tumorklassifikation genotypische Marker histomorphologischen Kriterien voranstellt.
Allgemeines zur MRT bei Gliomen
Die Magnetresonanztomografie (MRT) ist die grundlegende Bildgebungstechnologie für die Diagnose und Lokalisationsdiagnostik zerebraler Gliome. Sie dient der Erfassung der Tumorkategorie und ihrer möglichen Differenzialdiagnosen wie z.B. Hirnabszess, Lymphom oder Metastase (Abb. 1a). Die MRT-Untersuchung ist von vielen Faktoren abhängig, wie z.B. Feldstärke, MR-Sequenzen, Scannertyp, Schichtdicke und Bildkontrast. Standardisierte Untersuchungsprotokolle sollten neben nativen T1- und T2-gewichteten Sequenzen hochauflösende «Fluid attenuated inversion recovery»- (FLAIR) und KM-gestützte T1-gewichtetete 3D-Sequenzen bei jeweils identischer Feldstärke umfassen (Empfehlungen der «European Organisation for Research and Treatment of Cancer» [EORTC] und der «National Brain Tumor Society» [NBTS] für strukturelle MRT).1 Die Integration von T1-gewichteten 3D-Sequenzen spielt insbesondere für die quantitative volumetrische Bestimmung der Tumorgeometrie und Tumorprogression und die Anwendung der RANO-(«Response Assessment in Neuro-oncology»)-Kriterien eine wichtige Rolle.2
Limitationen der konventionellen MR-Bildgebung und der qualitativen Beurteilung der Therapieansprache umfassen ihre geringe Sensitivität bezüglich der Unterscheidung von echter Tumorprogression gegenüber therapieinduzierten Effekten und ihre Abhängigkeit von individuellen Untersuchern.3 Die Anwendungen von Methoden des sogenannten «advanced neuroimaging» zur Gewebstypisierung, wie MR-Spektroskopie, Perfusions- oder Diffusionsbildgebung, bieten diesbezüglich eine höhere Sensitivität und Spezifizität, sind gegenüber den Standardprotokollen aber kaum standardisiert. In einer Umfrage von insgesamt 220 Zentren in Europa gaben 85% der befragten Zentren an, Perfusionsbildgebung als Standard in der Gliombildgebung implementiert zu haben, wohingegen die MR-Spektroskopie nur in Einzelfällen und bei spezifischer Indikationsstellung zum Einsatz kommt, aber ebenfalls in ca. 80% der Zentren methodisch vorgehalten wird.4
Die Perfusionsbildgebung
Die Perfusionsbildgebung basiert mehrheitlich auf den sogenannten T2*-Effekten («dynamic susceptibility contrast», DSC), als alternative Methode stehen die T1-gewichteten («dynamic contrast enhanced», DCE) zur Verfügung. Die DCE-Perfusion bietet Vorteile bezüglich der Quantifizierung der Blut-Hirn-Schranken-Störung von Gliomen als Marker für den histopathologischen Malignitätsgrad, die DSC-Perfusion bezüglich der Erfassung des zerebralen Blutflusses und der Transitzeiten des Kontrastmittels im Tumor und in dessen Umgebungsödem.5 In einem systematischen Vergleich beider Methoden zeigten sich beide Methoden gleichwertig in der Unterscheidung zwischen Tumorgewebe und therapiebezogenen Effekten (Abb. 1b).6
Neuere Methoden zur Erfassung der Mikrozirkulation wie die «Intravoxel incohorent motion»(IVIM)-Perfusion oder das «arterial spin labeling» (ASL) ermöglichen die Erfassung der regionalen Durchblutungsveränderungen im Tumorgewebe ohne Kontrastmittelapplikation, werden bis heute jedoch mehrheitlich im Rahmen von Studien zur Vorhersage von Therapieansprache und für das Tumorgrading eingesetzt.7, 8
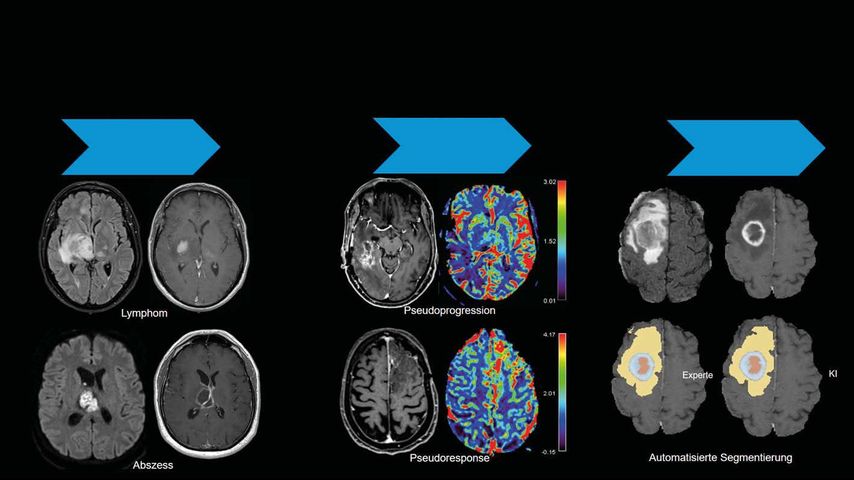
Abb. 1: MR-Bildgebung in der Neuroonkologie. a) Bildgebende Abgrenzung von höhermalignen Gliomen und ihrer Differenzialdiagnose wie z.B. Lymphomen und Abszessen in der Primärdiagnostik (Abszess: DWI-Restriktion und lineares Randenhancement, Lymphom: DWI/FLAIR-Signalanhebung und homogenes Enhancement). b) Abgrenzung von Pseudoprogression (rCBV ↓) und Pseudoresponse (rCBV ↑). c) Tumorvolumetrie eines Glioblastoms. Obere Reihe: FLAIR und T1-KM Sequenz. Untere Reihe: links: Segmentierung durch radiologischen Experten, rechts: Segmentierung durch den Deep-Learning-basierten Algorithmus «Deep SCAN 2020» (Dr. R. McKinley/Dr. R. Meier, Bern). Gelb: Tumorödem, hellblau: KM-aufnehmender Anteil, orange: zentrale Nekrose
Genetische Merkmale zur modernen Tumorklassifikation
IDH(Isocitrat-Dehydrogenase)-Status und 1p/19q-Kodeletion sind wichtige Aspekte der modernen Tumorklassifikation. IDH-Mutationen sind an der Gliomgenese beteiligt und kommen bei den meisten Gliomen der Grade 2 und 3 sowie bei sekundären Glioblastomen vor.9 Oligodendrogliale Tumoren werden anhand der 1p/19q-Kodeletion klassifiziert. Frontale und parietale Prädilektion, unscharfe Tumorbegrenzungen oder Kalzifikationen bei den 1p/19q-kodeletierten Gliomen oder temporale oder insuläre Prädilektion und homogenere Signalintensitäten der Tumormatrix erlauben eine erste Abschätzung der vorliegenden Tumorentität (Tab.1).10–13
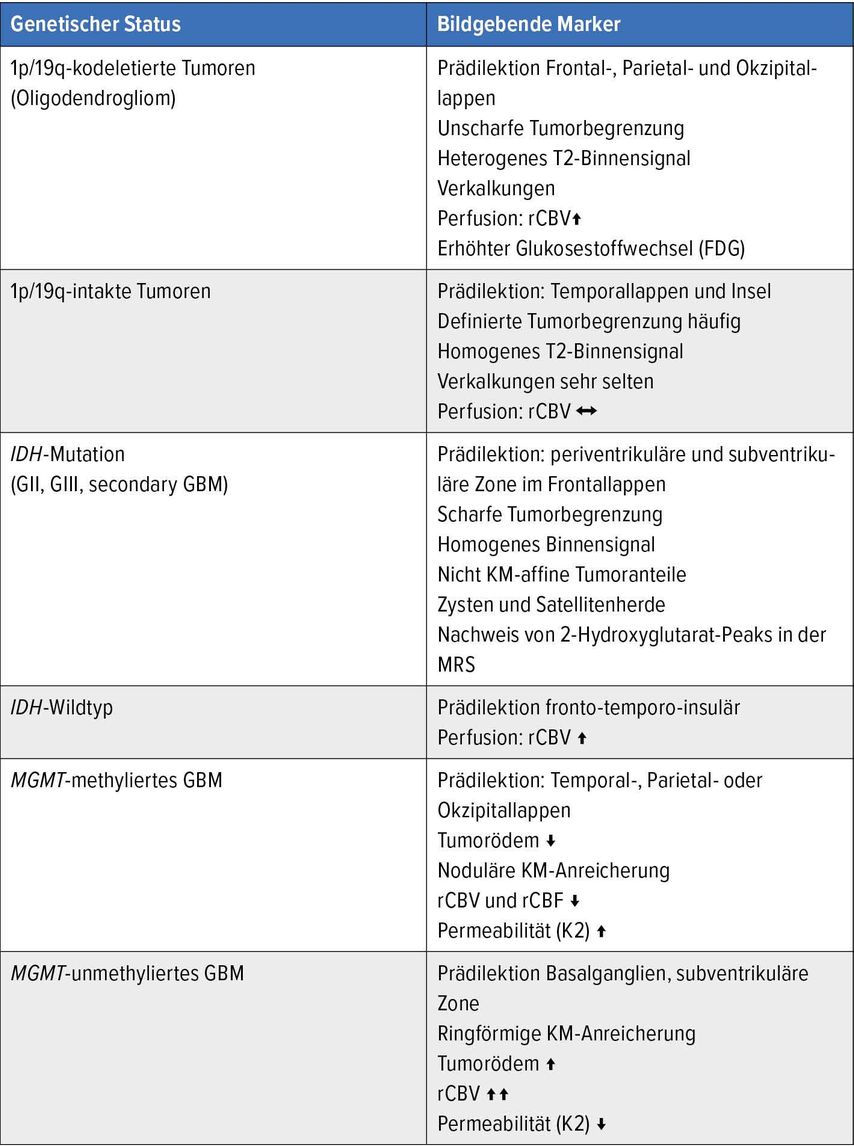
Tab. 1: Tumorklassifikation: bildgebende Marker nach genetischem Status. FDG: Fluorodeoxyglucose; GBM: Glioblastom; rCBF: «relative cerebral blood flow»; rCBV: «relative cerebral blood volume»
Die Magnetresonanzspektroskopie erlaubt neben einer Beurteilung des Malignitätsgrades anhand der Expression von Markern der neuronalen Signaltransmission, des Zellmembran-Metabolismus und von Zellnekrosen die indirekte Erfassung genetischer Marker durch Nachweis von 2-Hydroxyglutarat14 bei IDH-mutierten Tumoren.15–17 Die MR-Spektroskopie zeigt sich hierbei in der Abgrenzung zwischen IDH-Wildtyp und IDH-mutierten Gliomen sowohl der konventionellen wie auch der funktionellen Bildgebung überlegen.10, 18
APT-gewichtete Bildgebung
Die «amid proton transfer»(APT)-gewichtete Bildgebung («chemical exchange saturation transfer») ist ein derzeit noch weitgehend experimentelles MRT-Verfahren, dessen Bildkontrast auf dem chemischen Austausch zwischen Wasserstoffatomen und mobilen Protonen in Amiden, Aminen und Hydroxylgruppen beruht.19,20 Das Signal der Protonen von Peptidketten in Proteinen ist zu gering, um im normalen MRT gemessen werden zu können. Der Austausch zwischen Protein-Amid-Gruppen und umgebendem Wasser eröffnet eine Möglichkeit zur Messung dieser Protonen.
Ein potenzielles Einsatzfeld für diese Methode ist neben der Differenzierung des histopathologischen Malignitätsgrades und der Abgrenzung von therapieassoziierten Effekten die nicht invasive Unterscheidung von IDH-Wildtyp- und IDH-mutierten Gliomen und des MGMT(O-6-Methylguanin-DNA-Methyltransferase)-Methylierungsstatus.21–23 Die Entwicklung der Methode für klinische Anwendungen befindet sich derzeit in Vorbereitung.
Bildanalyse mittels künstlicher Intelligenz
Die Vorhersage des Genotyps anhand des bildgebenden Phänotyps ist auch Ziel von KI(künstliche Intelligenz)-basierten Bildanalysen. Ziel der KI-gestützten Bildanalyse ist es, Algorithmen so zu trainieren, dass sie die optimale Wiedergabe des genotypischen Profils oder einer Gewebsklasse vorhersagen.24 Mittels KI-Verfahren können Differenzierungen genetischer Mutationen25–27 und regionaler genetischer Heterogenität vorgenommen werden oder geeignete Subregionen für eine Biopsie28,29 oder eine personalisierte Strahlentherapie definiert werden.30 Darüber hinaus kann die automatisierte Tumorsegmentierung zwischen tumorassoziiertem Ödem, Tumornekrose und schrankengestörten Tumoranteilen unterscheiden (Abb.1c). So werden auf neuronalen Netzwerken basierende Expertensysteme (häufig mithilfe von Verfahren des «deep learning») bereits ergänzend zur visuellen Analyse für die Volumenberechnung von Gliomen herangezogen und berechnen das Tumorvolumen innerhalb weniger Sekunden mit einer vergleichbaren Fehlerrate wie medizinische Experten. 24,31 In einer retrospektiven Analyse in einem multizentrischen Datensatz aus einer prospektiven, randomisierten Phase-II- und -III-Studie (EORTC-26101) zeigte sich die automatisierte Bewertung des Tumoransprechens der zentralen RANO-Bewertung zur Vorhersage des Gesamtüberlebens deutlich überlegen.32
In Kombination mit sogenannten «radiomischen» Bildanalysen können Strukturen in Bilddaten erfasst werden, die charakteristische und prognostisch relevante Bildgebungsmerkmale in der Tumormatrix detektieren, welche deren Heterogenität auf molekularer Ebene repräsentieren.27 Hierzu werden Korrelationsprüfungen zwischen quantitativen Merkmalen wie z.B. Form, Signalverteilung und Texturen durchgeführt, um statistische Aussagen über den Malignitätsgrad, Therapieansprache und den Krankheitsverlauf machen zu können.33 Erste retrospektiv durchgeführte Studien belegen einen von klinischen und molekularen Markern unabhängigen Vorhersagewert für das progressionfreie Überleben und das Gesamtüberleben von Gliompatienten, sind jedoch derzeit noch nicht Teil klinischer Bildgebungsprotokolle.
Fazit
Die laufende Weiterentwicklung der neuroonkologischen Bildgebung zielt auf
die Abgrenzung therapieassoziierter Veränderungen von Tumorprogression,
die Identifikation bildgebender Korrelate genetischer Marker und
eine Prognoseabschätzung unter Therapie.
Hierzu kommen Verfahren des «advanced neuroimaging» und Verfahren der computerunterstützten, automatisierten Bildanalyse zum Einsatz. Aus diesen Weiterentwicklungen entstehen neue interdisziplinäre Herausforderungen und Berufsprofile, für deren Umsetzung eine enge Zusammenarbeit zwischen Neuropathologen, Ingenieuren, Bildinformatikexperten und (Neuro-)Radiologen unabdingbar ist.
Literatur:
1 Ellingson BM et al.: Neuro Oncol 2015; 17(9): 1188-98 2 Ellingson BM et al.: Neurotherapeutics 2017; 14(2): 307-20 3Young RJ et al.: Neurology 2011; 76(22): 1918-24 4 Thust SC et al.: Eur Radiol 2018; 28(8): 3306-17 5 Artzi M et al.: Neuroradiology 2015; 57(7): 671-8 6 Patel P et al.: Neuro Oncol 2017; 19(1): 118-27 7 Federau C et al.: AJNR Am J Neuroradiol 2014; 35(2): 256-62 8 Hashido T et al.: Sci Rep 2020; 10: 6121 9 Han S et al.: I Br J Cancer 2020; 122(11): 1580-9 10 Smits M et al.: Radiology 2017; 284(2): 316-31 11Ellingson BM et al.: AJNR Am J Neuroradiol 2013; 34(3): 533-40 12 Kickingereder P et al.: IDH mutation status is associated with a distinct hypoxia/angiogenesis transcriptome signature which is non-invasively predictable with rCBV imaging in human glioma.Sci Rep 2015; 5: 16238 13Suh CH et al.: AJNR Am J Neuroradiol 2018; 39(8): 1439-4514 Öz G et al.: Radiology 2014; 270(3): 658-79 15 Andronesi OC et al.: Sci Transl Med 2012; 4(116): 116ra4 16 Choi C et al.: Nat Med 2012; 18(4): 624-9 17 Zhou M et al.: Neuro Oncol 2018; 20(9): 1262-71 18 Suh CH et al.: Neuro Oncol 2018; 20(12): 1573-83 19 Zhou J, van Zijl PCM: Prog Nucl Magn Reson Spectrosc 2006; 48(2-3): 109-36 20 Zhou J et al.: Magn Reson Med 2003; 50(6): 1120-6 21 Jiang S et al.: Magn Reson Med 2017; 78(3): 1100-9 22 Kamimura K et al.: Jpn J Radiol 2019; 37(2): 109-16 23 Zhou Jet al.: J Magn Reson Imaging 2019; 50(2): 347-64 24 Rebsamen M et al.: Front Neurosci 2019; 13: 1182 25 Chang P et al.: AJNR Am J Neuroradiol 2018; 39(7): 1201-7 26 Eichinger P et al.: Sci Rep 2017; 7(1): 13396 27 Hu LS et al.: Neuro Oncol 2017; 19(1): 128-37 28 Chang PD et al.: AJNR Am J Neuroradiol 2017; 38(5): 890-8 29 Menze BH et al.: Inf Process Med Imaging 2011; 22: 735-47 30. Lipkova J et al.: IEEE Trans Med Imaging 2019; 38(8): 1875-84 31 Rebsamen M et al.: Front Neurol 2020; 11: 244 32 Kickingereder P et al.: Lancet Oncol 2019; 20(5): 728-40 33 Soni XN et al.: AJNR Am J Neuroradiol 2019; 40(6): 928-34
Das könnte Sie auch interessieren:
Erhaltungstherapie mit Atezolizumab nach adjuvanter Chemotherapie
Die zusätzliche adjuvante Gabe von Atezolizumab nach kompletter Resektion und adjuvanter Chemotherapie führte in der IMpower010-Studie zu einem signifikant verlängerten krankheitsfreien ...
Highlights zu Lymphomen
Assoc.Prof. Dr. Thomas Melchardt, PhD zu diesjährigen Highlights des ASCO und EHA im Bereich der Lymphome, darunter die Ergebnisse der Studien SHINE und ECHELON-1
Aktualisierte Ergebnisse für Blinatumomab bei neu diagnostizierten Patienten
Die Ergebnisse der D-ALBA-Studie bestätigen die Chemotherapie-freie Induktions- und Konsolidierungsstrategie bei erwachsenen Patienten mit Ph+ ALL. Mit einer 3-jährigen ...