Genetische Diagnostik in der hämatologischen Präzisionsmedizin
Autor:
Priv.-Doz. Dr. Gregor Hörmann, PhD
MLL Münchner Leukämielabor, München
E-Mail: gregor.hoermann@mll.com
Das Zusammenspiel von Verbesserungen der Diagnostik und der Therapie ist der Schlüssel zum Verständnis und zur Heilung von Leukämien und Lymphomen. Eine umfassende phänotypische und genetische Charakterisierung von Tumorproben erlaubt es uns, die Pathophysiologie der Erkrankungen zu verstehen, Achillesfersen der Tumorbiologie als therapeutische Targets zu identifizieren und spezifische Medikamente dagegen zu entwickeln.
Keypoints
-
Moderne Diagnostik ist ein Eckpfeiler des Patient:innenmanagements in der Hämatologie und ermöglicht die Diagnosestellung, die Prognosestratifizierung, die Identifikation von therapeutischen Targets und die Verlaufsuntersuchung bei Patient:innen mit Leukämien oder Lymphomen.
-
Die Integration unterschiedlicher diagnostischer Methoden ergibt ein präzises Bild der Erkrankung und ermöglicht ein personalisiertes Monitoring des Therapieansprechens.
-
„Next-generation sequencing“ (NGS) erlaubt eine umfassende molekulargenetische Untersuchung von indikationsspezifischen Genpanels bis hin zum gesamten Genom in der hämatologischen Diagnostik.
Ein durchschlagendes Therapieansprechen macht hochsensitive diagnostische Methoden notwendig, um die minimale Resterkrankung („minimal residual disease“, MRD) unter oder nach Therapie zu quantifizieren, beginnende Rezidive frühzeitig zu erkennen und das Management der Patient:innen mit Leukämien und Lymphomen anzuleiten. Die Hämatologie ist hierbei u.a. durch die gute diagnostische Verfügbarkeit von Blut das Vorbild der Onkologie geworden und längst im Zeitalter der Präzisionsmedizin und der personalisierten Medizin angekommen.
Präzisionsmedizin und personalisierte Medizin
Grundlage der Präzisionsmedizin ist die umfassende Charakterisierung der Tumorproben mit modernen diagnostischen Methoden inklusive der Identifizierung von therapeutischen Targets.1 Obwohl dabei unterschiedliche Techniken zum Einsatz kommen, sind die Fortschritte der Molekulargenetik, insbesondere die Methoden des „next-generation sequencing“ (NGS), eng mit dem Begriff der Präzisionsmedizin verknüpft; diese ermöglichen es, eine Vielzahl von potenziellen Zielstrukturen gleichzeitig zu untersuchen.
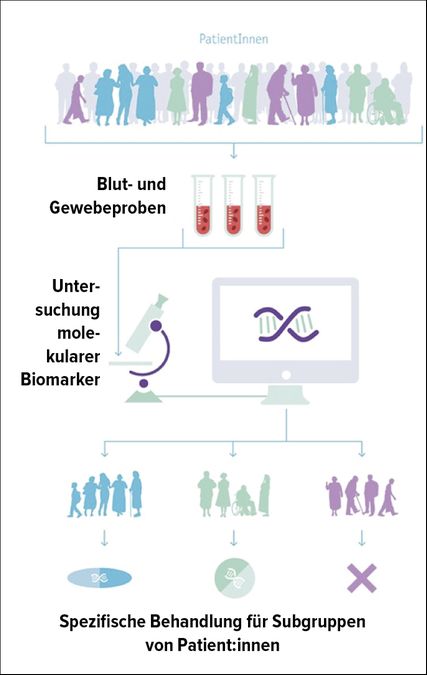
Abb. 1: Diagnostik als Grundlage der Präzisionsmedizin in der Hämatologie. Umfassende moderne Diagnostik erlaubt es, Patient:innen mit Leukämien oder Lymphomen in immer präzisere Subgruppen zu stratifizieren und deren Ansprechen auf bestimmte Therapieformen vorherzusagen
Auf Basis einer solchen Datengrundlage lassen sich die Patient:innen präzise in Subgruppen mit ähnlichen tumorbiologischen Eigenschaften einteilen, für welche ein homogener Krankheitsverlauf und ein ähnliches Ansprechen auf bestimmte (zielgerichtete) Therapien zu erwarten ist. Durch die Untersuchung prädiktiver Biomarker kann somit das therapeutische Management der Patient:innen optimiert werden (Abb. 1).
Der Begriff der personalisierten Medizin wird von manchen Autor:innen synonym zur Präzisionsmedizin verwendet, zumeist versteht man darunter jedoch im Kontext der Hämatologie, dass neben den tumorspezifischen Merkmalen auch patient:innenspezifische Eigenschaften stärker berücksichtigt werden.2
Diagnostische Methoden der Präzisionsmedizin in der Hämatologie
Neben allen modernen Techniken ist die Beurteilung des Blutbilds inklusive der traditionellen Zytomorphologie (Mikroskopie) des Blutes und des Knochenmarks das Rückgrat der hämatologischen Diagnostik, da sie in kurzer Zeit eine erste Einschätzung der zugrunde liegenden Pathologie erlaubt und das weitere diagnostische Vorgehen lenken kann.
Die Immunphänotypisierung oder FACS-Untersuchung charakterisiert die Oberflächenexpression von Antigenen auf neoplastischen Zellen und trägt so zur Identifizierung von Klonen, zur Liniencharakterisierung von Leukämien, aber auch zum Nachweis von Antigenen bei, die als Zielstrukturen für bestimmte therapeutische Antikörper und andere Arten der Immuntherapie dienen.
Die zytogenetischen Methoden der Chromosomenbänderungsanalyse und der Fluoreszenz-in-situ-Hybridisierung (FISH) weisen numerische und strukturelle Aberrationen von Chromosomen nach und sind beispielsweise geeignet, um die Translokation t(9;22)(q34;q11) als Korrelat der BCR::ABL1-Fusion oder die Deletion des Tumorsuppressorgens TP53 nachzuweisen.
Molekulargenetische Methoden umfassen eine Vielzahl von Techniken auf Basis der PCR und der Sequenzierung; sie sind geeignet, um Fusionsgene zu detektieren oder auf Einzelbasen-Niveau Punktmutationen in Genen zu identifizieren, wie zum Beispiel KIT D816V, JAK2 V617F oder auch diverse Mutationen im TP53-Gen.
Sowohl die Molekulargenetik als auch die Immunphänotypisierung sind geeignet, um nach Therapie eine geringe Anzahl residualer leukämischer Zellen hoch sensitiv nachzuweisen und so die MRD als wesentlichen Surrogat-Parameter für die Langzeitremission zu bestimmen.3
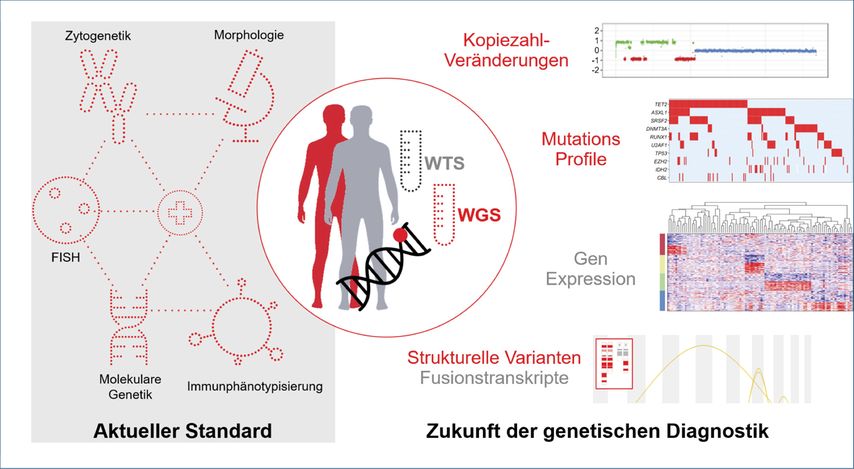
Aktuell ist es notwendig, mehrere komplementäre zyto- und molekulargenetische Methoden zu kombinieren und die genetische Diagnostik auf Basis der Zytomorphologie und/oder Immunphänotypisierung zu lenken. Die raschen Fortschritte der NGS-Verfahren machen es jedoch mittlerweile möglich, anstelle einzelner Gene oder mittelgroßer Genpanels auch das gesamte Genom („whole genome sequencing“, WGS) bzw. Transkriptom („whole transcriptome sequencing“, WTS) einer Tumorprobe zu untersuchen. WGS/WTS sind dazu geeignet, alle konventionellen Methoden der Zyto- und Molekulargenetik zu ersetzen und sämtliche Arten genetischer Aberrationen auf eine tumoragnostische Weise zu detektieren (Abb. 2).
Abb. 2: Genom- und Transkriptomsequenzierung als umfassende genetische Diagnostik der nahen Zukunft. „Whole genome sequencing“ (WGS, rot) und „whole transcriptome sequencing“ (WTS, grau) ergeben ein komplettes Bild der genetischen Veränderungen einer Leukämie bzw. eines Lymphoms und ersetzen konventionelle Methoden der Zyto- und Molekulargenetik
Obwohl der Aufwand für WGS/WTS derzeit noch höher ist als die Summe der konventionellen Verfahren, konnte bereits gezeigt werden, dass diese Methode in einem routinediagnostischen Setting anwendbar ist und einen Informationsgewinn bringt.4,5 Eine Translation von Forschung zur diagnostischen Anwendung steht damit unmittelbar bevor und kann bereits jetzt bei einzelnen unklaren Fällen von unmittelbarer Relevanz sein.
Beispiele von Anwendungen in der Hämatologie
Das beste Beispiel für diagnostische Werkzeuge der hämatologischen Präzisionsmedizin ist die chronische myeloische Leukämie (CML). Die CML ist durch den genetischen Nachweis der BCR::ABL1-Genfusion charakterisiert und das BCR::ABL1-Onkoprotein stellt auch ein direktes therapeutisches Target für Tyrosinkinaseinhibitoren (TKI) dar. Die TKI-Therapie hat die Behandlung der CML revolutioniert. Die Lebenserwartung von CML-Patient:innen unterscheidet sich heute kaum noch von derjenigen gesunder Personen.
Mittlerweile sind vier unterschiedliche TKI in der Erstlinientherapie der CML zugelassen, sodass auch das jeweilige Nebenwirkungsprofil und die Vorerkrankungen der Patient:innen in der Auswahl des TKI berücksichtigt werden können. Das Monitoring des Therapieansprechens ist von einer hochsensitiven PCR für BCR::ABL1 abhängig, die auch eine geringe MRD verlässlich quantifizieren kann und Therapieanpassungen erlaubt, bevor es zu einem hämatologischen Rezidiv kommt. Hierbei sind sekundäre Resistenzmutationen im BCR::ABL1-Gen eine wesentliche Ursache für die Therapieresistenz. Diese Resistenzmutationen können durch die Sequenzierung der Tyrosinkinasedomäne von BCR::ABL1 detektiert werden. Je nach Mutationsprofil kann dadurch das Ansprechen auf Zweit- und Drittgenerations-TKI vorhergesagt werden, was die molekulare Diagnostik zum Schlüssel für das therapeutische Management von CML-Patient:innen macht.6
Bei anderen hämatologischen Neoplasien ist der Grad der personalisierten Therapie noch nicht so hoch wie bei der CML, dennoch sind genetische Untersuchungsverfahren der Präzisionsmedizin mittlerweile Standard, um therapeutische Targets bei Patient:innen mit akuten Leukämien zu identifizieren und eine genaue Unterteilung der Diagnose vorzunehmen. Auch bei der chronischen lymphatischen Leukämie (CLL) ist die genetische Diagnostik zum Standard des therapeutischen Managements geworden. Hier ist insbesondere der Nachweis von Veränderungen in Genen, die eine Resistenz gegenüber bestimmten Therapien vermitteln (TP53-Mutationen und Deletionen für konventionelle Chemotherapie und BTK-Mutationen bzw. BCL2-Mutationen für die entsprechenden Inhibitoren), für die Therapieauswahl von unmittelbarer klinischer Relevanz.1
Resümee
Das Zusammenspiel von Diagnostik und Therapie ist die Stütze der hämatologischen Präzisionsmedizin, die Integration unterschiedlicher diagnostischer Methoden ergibt dabei ein präzises Bild der Erkrankung. Genetische Methoden ermöglichen aktuell eine exakte Diagnosestellung, eine genaue Prognosestratifizierung und eine prädiktive Aussage über das Ansprechen auf zielgerichtete Therapien.
Die diagnostischen Werkzeuge der hämatologischen Präzisionsmedizin bedingen eine intensive Kommunikation zwischen diagnostisch und klinisch tätigen Ärzt:innen (z.B. in Tumorboards), um in einem interdisziplinären Setting das Management von Patient:innen mit hämatologischen Neoplasien zu optimieren.
Literatur:
1 Schmidts I et al.: Precision medicine in therapy of non-solid cancer. Handb Exp Pharmacol 2022; online ahead of print: doi:10.1007/164_2022_608 2 Valent P et al.: Precision medicine in hematology 2021: definitions, tools, perspectives, and open questions. Hemasphere 2021; 5(3): e536 3 Heuser M et al.: 2021 update on MRD in acute myeloid leukemia: a consensus document from the European LeukemiaNet MRD Working Party. Blood 2021; 138(26): 2753-67 4 Duncavage EJ et al.: Genome sequencing as an alternative to cytogenetic analysis in myeloid cancers. NEngl J Med 2021; 384(10): 924-35 5 Haferlach T et al.: Genome sequencing in myeloid cancers. N Engl J Med 2021; 384(25): e106 6 Hochhaus A et al.: European LeukemiaNet 2020 recommendations for treating chronic myeloid leukemia. Leukemia 2020; 34(4): 966-84
Das könnte Sie auch interessieren:
Erhaltungstherapie mit Atezolizumab nach adjuvanter Chemotherapie
Die zusätzliche adjuvante Gabe von Atezolizumab nach kompletter Resektion und adjuvanter Chemotherapie führte in der IMpower010-Studie zu einem signifikant verlängerten krankheitsfreien ...
Highlights zu Lymphomen
Assoc.Prof. Dr. Thomas Melchardt, PhD zu diesjährigen Highlights des ASCO und EHA im Bereich der Lymphome, darunter die Ergebnisse der Studien SHINE und ECHELON-1
Aktualisierte Ergebnisse für Blinatumomab bei neu diagnostizierten Patienten
Die Ergebnisse der D-ALBA-Studie bestätigen die Chemotherapie-freie Induktions- und Konsolidierungsstrategie bei erwachsenen Patienten mit Ph+ ALL. Mit einer 3-jährigen ...