Neue Tests bei Infektionskrankheiten
Bericht:
Dr. Norbert Hasenöhrl
Vielen Dank für Ihr Interesse!
Einige Inhalte sind aufgrund rechtlicher Bestimmungen nur für registrierte Nutzer bzw. medizinisches Fachpersonal zugänglich.
Sie sind bereits registriert?
Loggen Sie sich mit Ihrem Universimed-Benutzerkonto ein:
Sie sind noch nicht registriert?
Registrieren Sie sich jetzt kostenlos auf universimed.com und erhalten Sie Zugang zu allen Artikeln, bewerten Sie Inhalte und speichern Sie interessante Beiträge in Ihrem persönlichen Bereich
zum späteren Lesen. Ihre Registrierung ist für alle Unversimed-Portale gültig. (inkl. allgemeineplus.at & med-Diplom.at)
Einen ganz wichtigen Schritt stellt die Beschleunigung der Empfindlichkeitstestung bakterieller Erreger dar, Stichwort: RAST. Aber auch neue Sequenzierungsmethoden könnten in den nächsten Jahren Eingang in die infektiologisch-klinische Praxis finden, wie Univ.-Prof. Dr. Robert Krause, Graz, bei der Jahrestagung der Österreichischen Gesellschaft für Innere Medizin (ÖGIM) erklärte.
Grundsätzlich lassen sich drei Arten von Tests bei Infektionskrankheiten unterscheiden“, erklärte Univ.-Prof. Dr. Robert Krause, Klinische Abteilung für Infektiologie, MedUni Graz. Methoden zum direkten Erregernachweis sind etwa Mikroskopie, Kultur, Antigentest, PCR, andere molekularbiologische Nachweise, PCR und Sequenzierung. „Natürlich können und sollten wir dann, wenn wir einen Erreger gefunden haben, auch eine Empfindlichkeitstestung durchführen“, fuhr Krause fort.
Ein indirekter Erregernachweis erfolgt über die Bestimmung von Antikörpern. Die dritte Methode ist die Messung der – allerdings natürlich unspezifischen – Interaktion zwischen Wirt und Pathogen, etwa durch Parameter wie die Blutsenkungsgeschwindigkeit, das CRP, das Procalcitonin, das Interleukin 6 oder andere Interleukine, aber auch suPAR („soluble urokinase plasminogen activator receptor) oder, derzeit noch experimentell, cDNA („complimentary DNA“). Bei suPAR handelt es sich um die lösliche Form eines zellständigen Rezeptors, die bei Immunaktivierung vermehrt nachgewiesen wird, bei cDNA um einen von einer reversen Transkriptase umgeschriebenen RNA-Strang, der zu diagnostischen Zwecken genützt werden kann.
RAST
RAST steht für „rapid antimicrobial susceptibility testing“. „Bisher hat es ja ca. einen Tag gedauert, bis wir von einem Keim, der auf einer Platte gewachsen ist, ein Antibiogramm machen konnten“, so der Infektiologe.
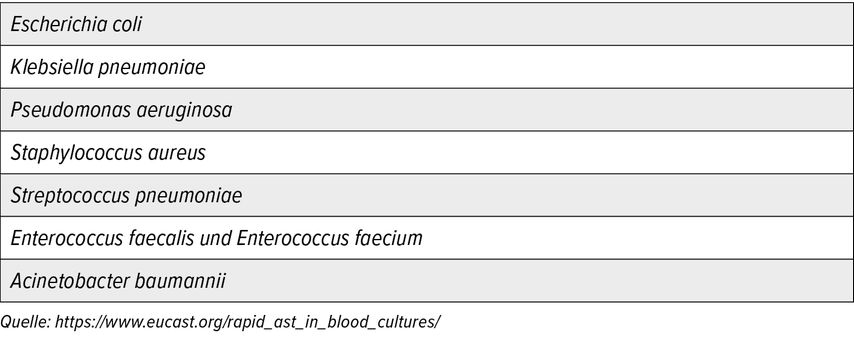
Das European Committee on Antimicrobial Susceptibility Testing, kurz EUCAST, hat aber 2019 eine Methode entwickelt, mit der es möglich ist, schon nach vier bis acht Stunden direkt aus positiven Blutkulturflaschen eine Empfindlichkeitstestung durchzuführen. Voraussetzung ist, dass die Identität des Erregers bereits bekannt ist, was mittels MALDI-TOF innerhalb weniger Minuten, bei positiver Subkultur in vier bis acht Stunden auf der Agarplatte durchgeführt werden kann. Die RAST-Methode ist für einige wichtige Erreger validiert, die in Tabelle 1 aufgelistet sind.
„Dazu gibt es eine eigene Breakpoint-Tabelle – die EUCAST betont, dass die regulären Breakpoints bei RAST nicht angewandt werden sollen“, warnte Krause.
Die Methode wurde in 55 Labors in Nord- und Südeuropa getestet und für verlässlich sowie für die tägliche Routine praktisch umsetzbar befunden.1 40 der beteiligten Labors lagen in Nord-, 15 in Südeuropa. Verwendet wurden klinische Blutkulturen mit E. coli, K. pneumoniae, P. aeruginosa, S. aureus und Pneumokokken. Die Resultate wurden nach vier, sechs und acht Stunden Inkubation abgelesen und mit Standard-Disk-Diffusionstests der EUCAST (16–20h) verglichen.1
Die Interpretierbarkeit der RAST-Ergebnisse stieg mit der Inkubationsdauer. Sie lag nach vier Stunden bei 70%, nach acht Stunden bei 85%. Die Fehlerraten nach acht Stunden lagen in Nordeuropa bei durchschnittlich 1,1%, in Südeuropa bei 3,3%.1 Eine falsche Empfindlichkeit lag bei 0,3% (Nordeuropa) bzw. 1,1% (Südeuropa) vor, eine falsche Resistenz bei 1,9% bzw. 2,8%. Insgesamt, so die Studienautoren, sei es bei 97% der positiven Blutkulturen mit der RAST-Methode möglich, nach vier bis sechs Stunden ein valides Resultat zu erhalten.1
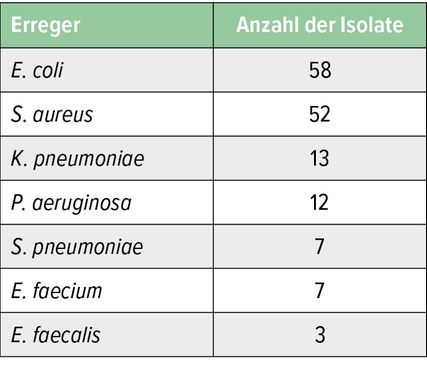
„Die Bedeutung dieser Methode liegt sicher darin, dass wir früher mit einer zielgerichteten Antibiotikatherapie beginnen können, was bekanntlich die Mortalität reduziert“, betonte der Experte. „In einer eigenen Studie haben wir uns dann angeschaut, was die Implementierung von RAST in der klinisch-infektiologischen Routine wirklich bringt“, fuhr Krause fort.2 152 Isolate wurden in die Studie aufgenommen, Tabelle 2 zeigt die Liste.
„Wir fanden nach vier Stunden bei 75,8% verwertbare Resultate, nach sechs Stunden bei 94,7%; dieses Resultat wurde dann nach acht Stunden auch nicht mehr besser“, berichtete Krause. Bei 134 Patienten wurde nach dem RAST eine infektiologische Konsultation durchgeführt. Bei fast der Hälfte (49%) führte der RAST zu einer Änderung der antimikrobiellen Therapie, bei weiteren 6% wurde dann erst eine antibiotische Therapie begonnen. Bei ca. 40% blieb die Behandlung unverändert, entweder weil sich der Erreger ohnehin als empfindlich erwies oder eine klinische Besserung eingetreten war.2
Eine Änderung oder ein Neubeginn der antimikrobiellen Therapie wurde am häufigsten bei Infektion mit S. aureus (45/50) und E. faecium (6/7) vorgenommen, weniger häufig bei den anderen getesteten Erregern.2 „Zudem konnten wir bei 80% der RAST-Patienten zusätzliche Maßnahmen empfehlen, wie etwa bildgebende Untersuchungen oder chirurgische Eingriffe“, ergänzte Krause.
Ein Vorteil bei der 30-Tages-Mortalität ergab sich durch die RAST-Anwendung hingegen nicht.2 „Es gibt aber schon ältere Daten aus unserer Gruppe, die zeigen, dass beiBakteriämien mit S. aureus ein infektiologisches Konsil erst ca. ab Tag 40 der Nachbeobachtung einen Unterschied für die Mortalität macht“, betonte der Infektiologe.3
„Ich denke, dass RAST die Mikrobiologie und die Zusammenarbeit zwischen Labor und Klinikern verändern und verbessern wird“, zeigte sich Krause überzeugt.
Neue Sequenzierungsmethoden
Es gibt eine Reihe von neuen Sequenzierungsmethoden, die unter anderem unter Begriffen wie „Amplicon-Sequenzierung“ oder „metagenomische Sequenzierung“ bekannt sind.4 „Amplicon-Sequenzierung bedeutet, dass man bestimmte, hochkonservierte Regionen der DNA von Bakterien oder Pilzen amplifiziert und mit entsprechenden Bibliotheken vergleicht, um eine Speziesidentifikation zu ermöglichen“, erläuterte Krause. Bei der metagenomischen Sequenzierung wird im Grunde alles sequenziert, was an Nukleinsäuren vorhanden ist (wobei RNA zuvor durch eine reverse Transkriptase in eine DNA umgeschrieben wird).4 „Hier wird dann durch hochkomplexe biostatistische Rechenmethoden ermittelt, welche Erreger vorliegen“, so der Experte.
Auch diese Methoden sind in der klinischen Praxis bereits verwendbar. Für Bakterien wird bei der Amplicon-Sequenzierung auf die DNA-Sequenz der sogenannten ribosomalen 16S-RNA abgezielt, bei Pilzen auf die sogenannten ITS-Sequenzen.
Hier zeigte eine Grazer Studie, dass die ITS-Sequenzierung bei 15% von 71 Patienten die antimykotische Therapie veränderte, während sie bei 27% die bereits laufende Therapie bestätigte und bei 48% die Entscheidung, nicht antimykotisch zu behandeln, unterstützte.5 „Dazu muss man sagen, dass wir hier unterschiedlichste Materialien verwendet haben, z.B. Lungengewebe, BAL, Blutproben, aber auch Hirngewebe, um fungale Erreger zu finden, die anders nicht zu identifizieren waren“, erklärte Krause. „Die ITS-Sequenzierung war also ein nützliches Zusatzinstrument zum bereits bestehenden diagnostischen Armamentarium für Pilzinfektionen.“
Die metagenomische Next-Generation-Sequenzierung (mNGS) wurde in Graz in einer Pilotstudie ebenfalls auf ihre Praxistauglichkeit überprüft, und zwar bei 42 Patienten mit hämatologischen malignen Erkrankungen und febriler Neutropenie (FN), die insgesamt 63 Episoden von FN erlebten.6 Während mittels Blutkulturen nur bei 12,7% der FN-Episoden ein Erreger zu finden war, war dies mittels mNGS bei 52,4%, nach Ausschluss viraler Erreger noch bei 38,1% der Fall – ein signifikanter Unterschied. Dazu ist zu sagen, dass 95% der Patienten zum Zeitpunkt der Blutabnahmen eine Antibiotikatherapie hatten.6
„Was uns diese Methode allerdings nicht liefert, ist die Interpretation, ob es sich bei den gefundenen Erregern auch wirklich um das kausale Agens für die Bakteriämie handelt oder nur um zufällig im Blut vorhandene DNA“, schränkte Krause ein. „Nebenbei bemerkt konnten alle im mNGS gefundenen Viren dann auch durch konventionelle PCR-Untersuchungen nachgewiesen werden.“
„Derzeit sind diese Methoden zwar noch sehr teuer und aufwendig, aber ich denke, dass sie bald Eingang in den klinischen Alltag finden werden“, sagte der Experte abschließend.
Quelle:
„Neue Tests bei Infektionskrankheiten“, Vortrag von Univ.-Prof. Dr. Robert Krause, Graz, im Rahmen der Jahrestagung der Österreichischen Gesellschaft für Innere Medizin (ÖGIM) am 23.9.2021
Literatur:
1 Åkerlund A et al.: J Antimicrob Chemother 2020; 75(11): 3230-8 2 Valentin T et al.: J Antimicrob Chemother 2021; 76(1): 233-8 3 Valentin T et al.: Clin Infect Dis 2014; 58(4): 599-600 4 Chiu CY, Miller SA: Nat Rev Genet 2019; 20(6): 341-55 5 Guenter S et al.: J Fungi (Basel) 2020; 6(2): 43 6 Schulz E et al.: ECCMID 2021; Abstract #3730
Das könnte Sie auch interessieren:
Diskriminierung von Menschen mit HIV in Deutschland und Österreich
Leider müssen Menschen, die mit HIV leben, auch im Jahr 2025 noch mit Schlechterbehandlung und Ablehnung leben – überwiegend in Hinblick auf Leistungen im Gesundheitsbereich. Die ...
Update EACS-Guidelines
Im schottischen Glasgow fand im November 2024 bereits zum 31. Mal der KongressHIV Drug Therapy Glasgow, kurz HIVGlasgow, statt. Eines der Highlights des Kongresses war die Vorstellung ...
Best of CROI 2025
Im März 2025 fand in San Francisco die 32. Conference on Retroviruses and Opportunistic Infections (CROI) statt. Wie gewohnt nahmen zahlreiche Expert:innen teil, um diverse ...