Multimodale Diagnostik und digitale Pathologie
Bericht:
Torsten U. Banisch, PhD
Sie sind bereits registriert?
Loggen Sie sich mit Ihrem Universimed-Benutzerkonto ein:
Sie sind noch nicht registriert?
Registrieren Sie sich jetzt kostenlos auf universimed.com und erhalten Sie Zugang zu allen Artikeln, bewerten Sie Inhalte und speichern Sie interessante Beiträge in Ihrem persönlichen Bereich
zum späteren Lesen. Ihre Registrierung ist für alle Unversimed-Portale gültig. (inkl. allgemeineplus.at & med-Diplom.at)
Beim Frühjahrs-Meeting der Österreichischen Gesellschaft für Klinische Pathologie und Molekularpathologie in Wien wurden neue Erkenntnisse zu Lungenkrebs, gastrointestinalen und gynäkologischen Karzinomen präsentiert. Unter anderem wurden die Schwierigkeiten in der Frühdiagnose und die Notwendigkeit einer multimodalen Diagnostik und einer multidisziplinären Expertise bei der Behandlungsauswahl diskutiert.
Session: Lungenkrebs
Aufkommende Marker für die zielgerichtete Behandlung von NSCLC
Univ.-Prof. Dr. Reinhard Büttner vom Universitätsklinikum Köln erklärte, dass die Grundlage einer maßgeschneiderten Therapieauswahl die Vielzahl bekannter molekularer Marker ist. Um einen andauernden Behandlungserfolg zu ermöglichen, müssen Patient:innen über die gesamte Behandlungsdauer hinweg analysiert werden, da sich molekulare Muster verschieben und Therapien angepasst werden sollten. Um dies zu gewährleisten, müssen rare Mutationen, deren Zusammenspiel und auftretende Resistenzmechanismen charakterisiert werden.1
Als Pilotprojekt hat das nationale Netzwerk Genomische Medizin (nNGM) eine präklinische, prognostische Plattform ins Leben gerufen, die unbekannte oder schlecht verstandene Mutationen im Vorfeld in vitro testet und Behandlungsempfehlungen erarbeitet. Dieser Forschungsansatz konnte unter anderem ermitteln, wie bestimmte Mutationen die Wirkungsweise von Wirkstoffen verändern.2 Ein prominentes Beispiel sind die „Flat type“-Tyrosinkinasen: Eine Struktur-Funktions-Analyse an atypischen EGFR-Mutationen, die 10–30% der EGFR-Mutationen ausmachen, konnte zeigen, dass vor allem Exon-20-Insertionen resistent gegenüber Tyrosinkinase-Inhibitoren (TKI) der dritten Generation waren. Bei dieser Resistenz blockieren Mutationen in der ATP-Bindestelle oder in PAAC die Bindung der TKI. Jedoch sind diese Mutationen sensitiv gegenüber TKI der zweiten Generation.3,4
Auch MET-Fusionen, die bei 0,1% der Patient:innen auftreten, verändern das Ansprechen auf Standardtherapien. Dank molekularer Charakterisierung konnten auch hier die Resistenzmechanismen identifiziert werden und es konnte eine Sensitivität gegenüber TKI der dritten Generation nachgewiesen werden, wodurch ein langes, dauerhaftes Ansprechen erreicht werden konnte.5
KEAP1-Mutationen sind mit einer schlechten Prognose assoziiert, da Resistenzen gegenüber Immuntherapien und Platin vorliegen. Diese treten auf, da das KEAP1-Zielgen NRF2 nicht mehr gebunden und degradiert werden kann.6
Der neue, vielversprechende NRF2-Inhibitor R16 bindet selektiv mutiertes KEAP1 und rekonstituiert so die Bindestelle für NRF2.7 In der Präklinik wurde bereits gezeigt, dass NRF2 hierdurch degradiert werden kann, zudem erlangen behandelte Zellen in vitro erneut eine Sensitivität gegenüber Platin und TKI.7
Die Studie eröffnet somit einen neuen Therapieansatz: die gezielte Ausschaltung von Resistenzmechanismen und anschließende Anwendung von TKI und Immuntherapien.
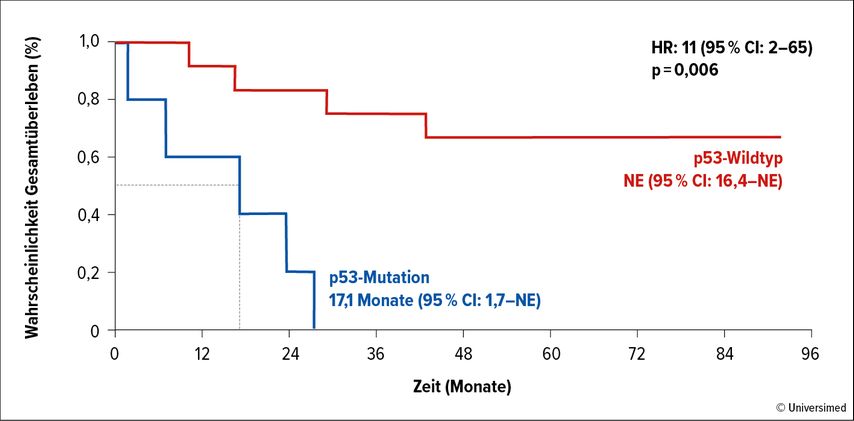
Eine weitere Schwierigkeit in der Behandlung sind Komutationen. Patient:innen mit ROS-Mutationen aus der ersten europäischen Studie zu Crizotinib wurden rezent nach einem medianen Nachbeobachtungszeitraum von 81,4 Monaten re-analysiert.8,9 Die Patient:innen teilten sich in zwei Gruppen auf: Jene mit ROS-Fusion und Wildtyp p53 hatten noch immer nicht 50% Gesamtüberleben (OS) erreicht. Patient:innen mit einer p53-Komutation hingegen hatten ein medianes OS von nur 17,1 Monaten (Abb. 1). Somit haben Komutationen, gerade p53, einen großen Einfluss auf das Ergebnis.9 Vergleichbares konnte auch bei Patient:innen mit ALK-Fusionen oder EGFR-Mutationen mit oder ohne p53-Komutationen gezeigt werden.10,11
Abb. 1: Gesamtüberleben (OS) unter Crizotinib bei NSCLC mit oder ohne p53-Komutation (modifiziert nach Michels S et al.)9
Diese Unterschiede können durch die Vielzahl zusätzlicher Mutationen in p53-mutierten Genomen erklärt werden.12 Die damit bestehende hohe Tumorheterogenität ist nur schwer durch einen einzelnen TKI zu kontrollieren. Eine Kombinationstherapie aus mehreren TKI und einer Chemotherapie ist daher ein vielversprechenderer Ansatz.
Die angeführten Beispiele verdeutlichen den klinischen Nutzen, unklare/komplexe Genotypen intensiv auszutesten und experimentell zu evaluieren. Ansätze wie das nNGM-Modellsystem, das multidisziplinäre Expertisen integriert, können helfen, personalisierte Behandlungen für Patient:innen zu ermitteln.
Session: Gastrointestinale Karzinome
Vorläuferläsionen des ösophagealen Plattenepithelkarzinoms
Die Vorläufer des ösophagealen Plattenepithelkarzinoms (SCC) sind zwar leicht zu definieren, aber schwer zu diagnostizieren – mit dieser Aussage eröffnete Prof. Dr. Nina Zidar (Institut für Pathologie, Ljubljana, Slowenien) ihren Vortrag. Dies liegt an der Schnittmenge mit auftretenden Atypien und der schrittweisen Tumorentwicklung im Ösophagus.13
Zu den Vorläuferläsionen zählen die intraepithelialen Neoplasien niedrigen (LG) und hohen Grades (HG) und das Vorhandensein von atypischen Zellen, die auf das Epithel beschränkt sind. Gerade in der Diagnostik ist hierbei wichtig zu beachten, dass Atypien, wie reaktive/regenerative Atypien und solche, die nach einer Chemoradiotherapie auftreten, fälschlicherweise als Vorläuferläsion identifiziert werden können.
Durch Multi-Region-Whole-Genome-Sequenzierungen konnten Vorläuferläsionen und SCC charakterisiert werden, wobei die komplette Inaktivität von p53 eine Grundlage für die Entwicklung des SCC darstellt.14 Auch die Anzahl signifikant unterschiedlich exprimierter Gene steigt von LG (63) zu HG (826) und SCC (1298) stetig an. Die Proteine TAGLN2 und CRNN sind hierbei Indikatoren für das Risiko für einen Progress zum SCC. Diese Daten zeigten weiters, dass der neoplastische Klon genetisch schon frühzeitig vorhanden ist, es bleibt jedoch ungeklärt, wie eine Dysplasie in ein invasives SCC übergeht.15
15–24% der LG-Dysplasien und 30–74% der HG-Dysplasien entwickeln sich in einem Zeitraum von 8–13 Jahren zu einem SCC, auch das Risiko für Dysplasien und SCC in anderen Organen ist erhöht.16 Die derzeitigen Therapieoptionen sind die endoskopische Biopsie, die Resektion der Mukosa oder eine Dissektion der Submukosa.13
Gastrische Dysplasien und Frühstadien des Magenkarzinoms
Dr. Katerˇina Kamarádová (Universität Hradec Králové, Tschechische Republik) wies in ihrem Vortrag darauf hin, dass die Mehrheit der Vorläufer des Magenkarzinoms sowie der Dysplasien spontan auftreten, zumeist in einem Setting einer chronischen Entzündung, einer intestinalen Metaplasie, einer peptischen Ulkuserkrankung oder nichtneoplastischer Polypen, die Vorstufen sein können. Frühstadien sind auf die Mukosa und Submukosa limitiert, wohingegen sich Magenkarzinome histologisch durch eine Invasion in die Lamina propria oder über die Submukosa hinaus ausweisen.17
Eine Behandlung von Patient:innen mit LG-Dysplasie und solchen mit HG-Dysplasie oder Magenkarzinom im Frühstadium bei keinem oder minimalem Risiko für Lymphknotenmetastasen sollte mittels endoskopischer Resektion erfolgen.18
Diese kann als endoskopische Piecemeal-Mukosaresektion (pEMR) oder endoskopische Submukosa-Dissektion (ESD) durchgeführt werden. Beide Methoden haben Vor- und Nachteile. Die pEMR hat kürzere Operationszeiten und ein niedriges Risiko für eine Perforation, die ESD hingegen weist eine hohe Rate an Komplettresektionen und ein geringeres Wiederauftreten auf.18 Eine Resektion sollte im Idealfall die Diagnose bestätigen, das Staging bei invasiven Karzinomen ermöglichen, Risikofaktoren für Lymphknotenmetastasen oder eine Resterkrankung aufzeigen und Aufschluss über die Heilbarkeit oder weitere Behandlungen geben, inklusive chirurgischer Eingriffe.
Session: Gynäkologische Karzinome
Molekulare Klassifizierung von Endometriumkarzinomen
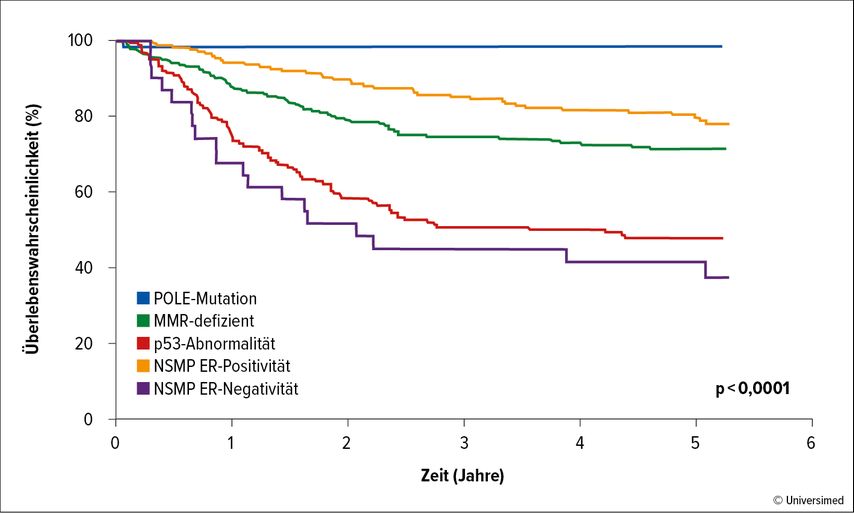
Mittels Next-Generation-Sequenzierungen können Endometriumkarzinome in molekulare Untergruppen eingeteilt werden, die zudem klare Prognosen erlauben: Tumoren mit POLE-Mutationen (ultramutierte Tumoren) haben generell ein positiveres klinisches Ergebnis, hypermutierte MMR-defiziente Tumoren mit MSI haben gute bis intermediäre Prognosen. Tumoren ohne spezielles molekulares Profil (NSMP), die Anomalien in geringer Kopienzahl aufweisen, haben ebenfalls gute bis intermediäre Prognosen, Tumoren mit hoher Kopienzahl von p53-Mutationen und seröser Histologie hingegen haben schlechte Outcomes, erklärte Prim. Univ.-Prof. Dr. Sigurd Lax (Landeskrankenhaus Graz).19
3,5% der Endometriumkarzinome weisen multiple Klassifizierungsmutationen auf, mit MMRd+p53 (60%), POLE+p53 (29%) und POLE+MMRd+p53 (11%). Interessanterweise ist die Prognose dieser Tumoren signifikant besser als die von p53-Tumoren, was vermuten lässt, dass p53 hier eine Passenger-Mutation ohne größeren klinischen Einfluss darstellt.20
Als zusätzliche Tumormarker sind L1CAM-Mutationen zu nennen, die die Prognose von NSMP-Karzinomen verschlechtern, zudem begünstigt ER-Positivität die Prognose von Hochrisiko-NSMP, ER-Negativität verschlechtert sie (Abb.2).21
Abb. 2: Molekulare Klassifikation und Prognose von Endometriumkarzinomen (modifiziert nach Vermin L et al.)21
Es gibt jedoch auch Veränderungen, die bisher noch keinen Einfluss auf das Vorgehen in der Praxis haben: Bei NSMP-Tumoren sind Veränderungen in PTEN/PI3KR1 begünstigend, Mutationen in PI3KCA/AKT1 und Amplifikationen in Chromosom 1q sind prognostisch schlechter.22
Diese molekularen Subtypen haben nicht nur eine prognostische Kraft, sondern diktieren auch Therapieoptionen. Die PORTEC-3-Studie konnte zeigen, dass Patient:innen mit p53-Mutationen einen signifikanten Nutzen von einer Kombination aus Chemo- und Radiotherapie gegenüber alleiniger Radiotherapie hatten. Ähnliche, aber weniger signifikante Beobachtungen wurden auch bei NSMP-Karzinomen gemacht.23 Diese Beobachtung wurde durch die Studien PORTEC-1 und PORTEC-2 gestützt, in denen gezeigt wurde, dass die molekulare Klasse des Tumors das Ansprechen auf eine Radiotherapie bei Endometriumkarzinomen im frühen Stadium prognostiziert.24
In Studien zu Folgetherapien, wie der Studie KEYNOTE-158, konnte gezeigt werden, dass MSI-H/dMMR-Tumoren im fortgeschrittenen Stadium gut auf Anti-PD-1-Therapien wie Pembrolizumab ansprechen.25 Kombinationstherapien aus Pembrolizumab und Lenvatinib konnten bei fortgeschrittenem, metastasiertem Endometriumkarzinom nach platinhaltiger Chemotherapie verbesserte PFS- und OS-Daten gegenüber alleiniger Chemotherapie ermöglichen, unabhängig vom MMR-Status.26
Aufgrund dieser Fortschritte werden aktuell die Richtlinien zur Behandlung des Endometriumkarzinoms überarbeitet und kontrovers diskutiert. Dies umfasst neue Kategorien, histologische Parameter und molekulare Daten.27,28
Digitalisierung und AI-augmentierte Diagnostik in der Pathologie
Zum Abschluss des Meetings gaben Dr. Michael Roehrl (Beth Israel Deaconess Medical Center, USA) und Dr. Heimo Müller (Medizinische Universität Graz) einen Ausblick auf die Zukunft der Pathologie.
Die digitale, vernetzte und multimodale Pathologie kann dabei helfen, Resistenzmechanismen zu erkennen und erfolgreiche Therapien aufzuzeigen. Schon jetzt können Slides semiautomatisch erstellt, über Remote-Mikroskopie digital komplett erfasst und durch Algorithmen ausgewertet werden. Diese Automatisierungen gehen auch mit einer Standardisierung in den Abläufen einher, was wiederum Vorteile für die Prognostik hat. Auch die Vorzüge von immer und überall abrufbaren digitalen Slide-Archiven sind wegweisend.
Mittels digitaler Pathologie wurde unter anderem bereits die Quantifizierung von Zelldichten an 3539 Biopsien von Patient:innen mit kolorektalem Karzinom automatisch vorgenommen. Diese Datenmenge lieferte einen Score, der als Prädiktor für die Zeit bis zum Progress, das erkrankungsfreie Überleben und das OS dient und dessen Aussagekraft durch zwei rezente klinische Studien belegt wurde.29,30
Auch die Integration von digitaler Pathologie, Transkriptomik und Genetik kann Aufschlüsse und Vorhersagen für bestmögliche Behandlungen geben. Am Beispiel des kolorektalen Adenokarzinoms wurde rezent gezeigt, wie solch ein Multiomik-Ansatz die metastatische Progression dieser Tumoren aufdecken kann.31
Logische Weiterentwicklungen sind in der computergestützten Pathologie zu sehen, in der AI-generierte Algorithmen bei der Datenerhebung und Analyse eine zentrale Rolle spielen werden – bis hin zur augmentierten Pathologie.32
Quelle:
Frühjahrs-Meeting der Österreichischen Gesellschaft für Klinische Pathologie und Molekularpathologie, 7.–9. März, Wien
Literatur:
1 Guo H et al.: Cells 2022; 11(20): 3200 2 Nationales Netzwerk Genomische Medizin (nNGM) Lungenkrebs. Online unter https://nngm.de/ 3 Robichaux JP et al.: Nature 2021; 597: 732-7 4 Janning M et al.: Ann Oncol 2022; 33(6): 602-15 5 Riedel R et al.: J Thorac Oncol 2024; 19(1): 160-5 6 Saleh MM et al.: J Thorac Onco 2022; 17: 76-88 7 Aboulkassim T et al.: Cell Rep 2023; 42: 113104 8 Michels S et al.: JThorac Oncol 2019; 14: 1266-76 9 Michels S et al.: ESMO Open 2024; 9(2): 102237 10 Janning M et al.: Ann Oncol 2022; 33(6): 602-15 11 Parikh K et al.: J Clin Oncol 2023; 41(Suppl 16): Abstr. #9029 12 Alidousty C et al.: J Thorac Dis 2019; 11: S3-S8 13 Stahl M et al.: OeGHO-Leitlinie, Stand Juni 2023 14 Chen XX et al.: Nat Commun 2017; 8(1): 524 15 Liu X et al.: Nat Commun 2023; 14: 4779 16 Van Tilburg L et al.: Dis Esophagus 2023; 36(12): doad045 17 Kim JM et al.: Cancer Res Treat 2019; 51(4): 1568-77 18 Hoffman A et al.: Visc Med 2021; 37(5): 358-71 19 Concin N et al.: Int J Gynecol Cancer 2021; 31(1): 12-39 20 León-Castillo A et al.: J Pathol 2020; 250(3): 312-22 21 Vermin L et al.: Brit J Cancer 2023; 128(7): 1360-8 22 Tabata J et al.: Gynecol Obstet Clin Med 2023; 3(1): 7-17 23 León-Castillo A et al.: J Clin Oncol 2020; 38(29): 3388-97 24 Horeweg N et al.: J Clin Oncol 2023; 41(27): 4369-80 25 Marabelle A et al.: J Clin Oncol 2020; 38(1): 1-10 26 Makker V et al.: N Engl J Med 2022; 386(5): 437-48 27 Berek JS et al.: Int J Gynaecol Obstet 2023; 162(2): 383-94 28 McCluggage WG et al.: Int J Gene Cancer 2024; 34: 138-43 29 Pagès F et al.: Lancet 2018; 391(10135): 2128-213 30 Ascierto PA et al.: Oncoimmunology 2020; 9(1): 1826132 31 Tanaka A et al.: Cell Rep 2024; 43(2): 113810 32 Plass M et al.: IEEE Comput Graph Appl 2022; 42(6): 47-57
Das könnte Sie auch interessieren:
Highlights zu Lymphomen
Assoc.Prof. Dr. Thomas Melchardt, PhD zu diesjährigen Highlights des ASCO und EHA im Bereich der Lymphome, darunter die Ergebnisse der Studien SHINE und ECHELON-1
ASH 2020 – Highlights zu den aggressiven Lymphomen
Highlight-Themen der virtuellen ASH-Jahrestagung im Dezember 2020 waren an erster Stelle die Immunonkologika in all ihren Variationen, aber auch Beispiele für innovative Sequenztherapien ...
Aktualisierte Ergebnisse für Blinatumomab bei neu diagnostizierten Patienten
Die Ergebnisse der D-ALBA-Studie bestätigen die Chemotherapie-freie Induktions- und Konsolidierungsstrategie bei erwachsenen Patienten mit Ph+ ALL. Mit einer 3-jährigen ...