Die Zukunft hat schon begonnen
Institut für Pathologie<br> Medizinische Universität Wien<br> E-Mail: leonhard.muellauer@meduniwien.ac.at
Klinisches Institut für Pathologie<br> Medizinische Universität Wien<br> E-Mail: ana-iris.schiefer@meduniwien.ac.at
Die ersten Institute für Pathologie wurden in der ersten Hälfte des 19.Jahrhunderts gegründet. Seitdem hat sich vieles getan. Die molekulare Pathologie hat sich als tragende Säule der klinischen Pathologie etabliert. Außerdem konnten bahnbrechende technologische Neuerungen wie das „Next-Generation Sequencing“ etabliert werden. Mit der fortschreitenden Digitalisierung werden Arbeitsprozesse weiter vereinfacht und standardisiert werden. Im Folgenden sollen die wichtigsten Entwicklungen der Pathologie mit Fokus auf die letzten 20 Jahre kurz dargestellt werden.
Keypoints
-
Die Pathologie des 21. Jahrhunderts integriert Morphologie und Genetik.
-
Die Pathologie identifiziert Krankheitsmechanismen und ermöglicht damit zunehmend eine auf tumorspezifische molekulare Aberrationen ausgerichtete Therapie (sog. „Präzisionsmedizin“).
-
Die Pathologie des 21. Jahrhunderts wird digital, „machine learning“ und „artificial intelligence“ gehören zum neuen Vokabular.
Die klinische Pathologie entwickelte sich aus dem Studium der makroskopischen Organ- und Gewebeveränderungen bei Autopsien.1 Die ersten Institute für Pathologie entstanden in der ersten Hälfte des 19. Jahrhunderts. Die Implementierung der Lichtmikroskopie in der zweiten Hälfte des 19. Jahrhunderts revolutionierte das Fach. Im 20. Jahrhundert wurden histologische Färbetechniken verfeinert, die Immunhistologie und In-situ-Hybridsierungstechniken etabliert und einzelne molekularpathologische Untersuchungen eingeführt. Die ersten Jahrzehnte des 21. Jahrhunderts waren hingegen geprägt von einer Expansion der molekularen Pathologie, einer noch stärkeren Integration der Pathologie in die klinischen Entscheidungsfindungen und dem Aufkeimen einer digitalen Transformation der Befundung.

Abb. 1:Cover der 2001 erschienenen Klassifikation hämatopoetischer Neoplasien „Pathology & Genetics – Tumours of Haematopoietic and Lymphoid Tissues“
Die WHO-Klassifikation von Neoplasien integriert Morphologie und Genetik
Die Diagnose von Tumorerkrankungen ist der Schwerpunkt der klinischen Pathologie. Eine reproduzierbare und international vergleichbare Diagnostik braucht Standards für die Interpretation der Morphologie, immunhistologische Färbungen, molekulare Zusatzuntersuchungen und die Nomenklatur. Die Weltgesundheitsorganisation (WHO) und die Internationale Agentur für Krebsforschung (IARC) erstellen mit internationalen Experten eine Klassifikation von Tumoren, welche in einer Serie von Referenzbüchern (sogenannten „blue books“) publiziert wird, regelmäßigüberarbeitet wirdund derzeit in 12 Bänden vorliegt, welche online abgerufen werden können (https://publications.iarc.fr/Book-And-Report-Series/Who-Classification-Of-Tumours).
Im Jahr 2001 erfolgte mit der Herausgabe der Klassifikation von hämatopoetischen Neoplasien mit dem Titel „Pathology & Genetics– Tumours of Haematopoietic and Lymphoid Tissues“ (Abb. 1) ein Paradigmenwechsel durch die Einbindung genetischer Befunde in die Tumorklassifikation. Dieser Weg wurde auch bei den Klassifikationen anderer Tumorentitäten weiterverfolgt und ausgebaut.
Die Pathologie wurde in den letzten Jahrzehnten noch stärkerin die Klinik integriert, insbesondere durch die Entwicklung von zielgerichteten Therapien und die damit verbundene Notwendigkeit der Bestimmung prädiktiver Biomarker. Ferner wurden für verschiedene Fachgebiete integrierende und immer die Pathologie einbeziehende Tumorboards etabliert, wobei diese Entwicklung durch das Entstehen von „Comprehensive Cancer Centers“ beschleunigt wurde.
Bezeichnend für die ausgerufene Ära der „Präzisionsmedizin“ ist auch die Etablierung „molekularer Tumorboards“, in welchen molekularpathologische Befunde, vor allem bei therapieresistenten Patienten, diskutiert werden.
Die molekulare Pathologie wird zu einer Säule der klinischen Pathologie
Die am stärksten wahrgenommene Veränderung der beiden letzten Jahrzehnte war die Expansion der molekularen Pathologie, wobei die Weiterentwicklung allerdings vorwiegend die Tumorpathologie und in geringerem Ausmaß auch die mikrobiologische Diagnostik betraf. Die vermehrte Integration molekularpathologischer Befunde in die klinische Pathologie führte in Österreich zu einer Änderung der Fachbezeichnung in „klinische Pathologie und Molekularpathologie“. Die Entstehung von Tumorerkrankungen als Folge genetischer Veränderungen wurde in den letzten beiden Jahrzehnten des 20. Jahrhunderts eindrucksvoll offengelegt und die Identifikation von Onkogenen und Tumorsuppressorgenen waren Meilensteine in der Identifikation kausaler molekularer Ursachen der Tumorpathogenese (Abb. 2).
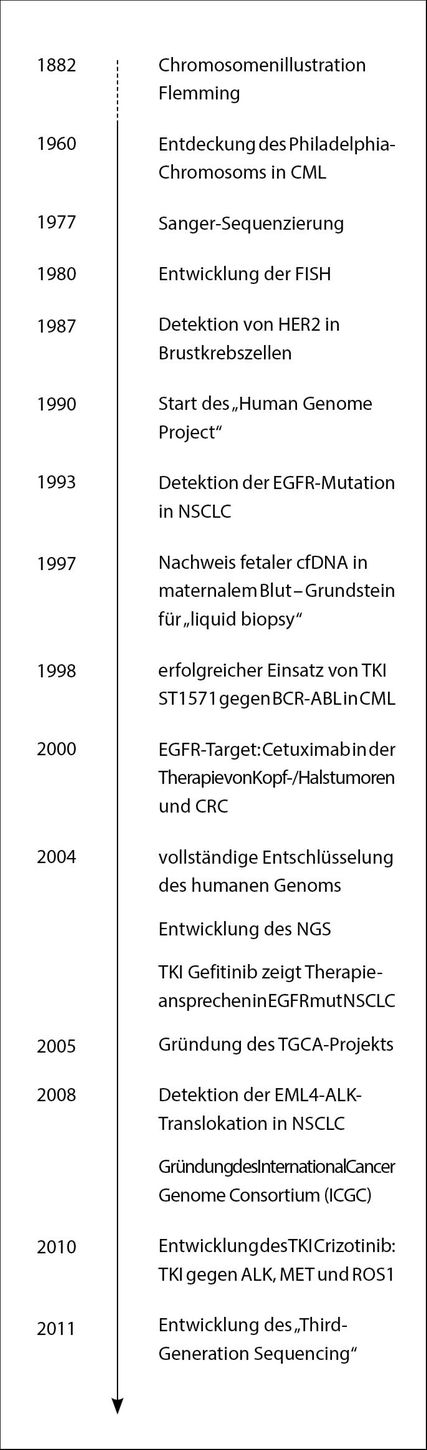
Abb. 2:Meilensteine der molekularen Pathologie. ALK: anaplastische Lymphomkinase; CML:chronische myeloische Leukämie; CRC:kolorektales Karzinom; FISH: Fluoreszenz-in-situ-Hybridisierung; NSCLC: nichtkleinzelliges Lungenkarzinom; TKI: Tyrosinkinase-Inhibitoren
Im 21. Jahrhundert erfolgte eine vermehrte Translation der Erkenntnisse der Krebsforschung in die Klinik. Die ersten historischen Beispiele für die Notwendigkeit der molekularpathologischen Bestimmung von Biomarkern für zielgerichtete Therapien bei soliden Neoplasien sind die HER2(„human epidermal growth factor receptor 2“)-Amplifikation beim Mammakarzinom und die KRAS-Mutationsbestimmung beim kolorektalen Karzinom für eine Anti-HER2- beziehungsweise Anti-EGFR(„Epidermal growth factor receptor“)-Antikörpertherapie sowie die EGFR-Mutationsanalyse beim Adenokarzinom der Lunge für eine Therapie mit Tyrosinkinaseinhibitoren (Abb. 2).
Die molekularpathologischen Untersuchungen dienen aber nicht nur der Identifikation von Therapietargets oder Resistenzmechanismen, sondern auch der Diagnose und Klassifikation von Tumoren, welche durch bestimmte genetische Aberrationen charakterisiert sein können. Für die Entwicklung der molekularen Tumorpathologie waren internationale Großprojekte wie die Entschlüsselung des humanen Genoms, welches 2004 fertiggestellt wurde, der Aufbau von großen Datenbanken mit normalen Gensequenzen Tausender Probanden (z.B. ExAC, „1000 Genomes Project“) als Referenz für genetische Variabilität sowie Datenbanken mit genetischen Tumoraberrationen wie der „Catalogue of Somatic Mutations in Cancer“ (COSMIC) wichtig. Ferner ermöglichten Großprojekte wie das in den USA verankerte Projekt „The Cancer Genome Atlas/TCGA“ und das „International Cancer Genome Consortium“ (ICGC) eine umfangreiche genetische Charakterisierung von verschiedenen Tumorentitäten mit DNA-, RNA- und Epigenomanalysen.
Die molekulare Pathologie wurde aber nicht nur durch den Wissenszuwachs in der Krebsforschung angetrieben, sondern durch damit eng verflochtene technische Neuerungen, welche den Entdeckungen der Forschung meist vorausgehen und diese erst ermöglichen. Dabei ist die Entwicklung des „Next-Generation Sequencing“ (NGS), welches ab 2004 Einzug in Forschunglabore hielt und ab 2010 sukzessive in diagnostische Labore aufgenommen wurde, von zentraler Bedeutung. Das NGS ist eine Revolution in der DNA- und RNA- Sequenzierung.2 Bis dahin war die von Frederic Sanger 1977 publizierte Sequenziertechnik in Verwendung (Abb.2). Damit wurde auch erstmalig das humane Genom sequenziert, was circa 13 Jahre in Anspruch nahm und kolportierte 3 Milliarden US-Dollar kostete. Im Vergleich dazu ist mit den derzeit potentesten NGS-Geräten die Sequenzierung eines menschlichen Genoms in einer Woche zu Kosten von circa 1000 US Dollar möglich. Der wesentlichste Unterschied zwischen der Sanger-Technologie und NGS ist der Sequenzierdurchsatz. Mit Sanger-basierter Gel-oder Kapillarelektrophorese zur Auftrennung der Sequenzierprodukte waren in Abhängigkeit von der Gelgröße, beziehungsweise ab den 1990er-Jahren der Anzahl von Gelkapillaren pro Sequenziergerät, 1–96 DNA-Fragmente gleichzeitig analysierbar. Mit NGS-Geräten können im Gegensatz dazu Hunderte Millionen von DNA-Fragmenten gleichzeitig sequenziert werden, weshalb die Technologie auch als „massive parallel sequencing“ bezeichnet wird.
In den molekularpathologischen Laboren werden meist Genpanel sequenziert, welche wenige bis über 500 Gene umfassen und je nach Design und Größe den Nachweis von Punktmutationen, kleinen Insertionen und Deletionen, Genkopieveränderungen und Genfusionen sowie auch Aussagen über eine Tumormutationslast und Mikrosatelliteninstabilität ermöglichen. Exom- und Genomsequenzierungen werden in der Routinediagnostik derzeit kaum verwendet. Die Gründe dafür sind eine fehlende klinische Indikation und die hohen Kosten, insbesondere bei einer Gesamtgenomsequenzierung.
Die Analyse von Tumorgewebe wurde durch die sogenannte „Liquid Biopsy“ ergänzt (Abb. 2). Dabei werden genetische Aberrationen in frei in Körperflüssigkeiten, insbesondere Blutplasma, zirkulierender DNA („circulating free DNA“, cfDNA) nachgewiesen. Die cfDNA wird während des physiologischen Zellumsatzes und bei pathologischen Prozessen von absterbenden Zellen freigesetzt. Im Plasma sind unter physiologischen Bedingungen pro Milliliter nur 1ng bis 10ng cfDNA vorhanden.3 Bei Tumorerkrankungen ist häufig, je nach Tumorlast und Tumorentität, die Menge von cfDNA im Blut erhöht. Die Analyse von cfDNA ermöglicht die Detektion von Therapietargets, die Identifikation von Resistenzmechanismen, den Nachweis einer minimalen Resterkrankung und das Monitoring eines Therapieansprechens. Die vom Tumor freigesetzte DNA macht meist nur unter 1% der cfDNA aus. Zum Nachweis von Mutationen in der cfDNA sind daher hochsensitive Analysemethoden wie „Digital droplet“-Polymerase-Kettenreaktion (PCR) oder NGS-Methoden mit „molecular barcoding“ notwendig.
Die digitale Pathologie erscheint am Horizont
Die digitale Pathologie ermöglicht die Auswertung von eingescannten histologischen Schnitten am Computerbildschirm. Vorteile im Vergleich zur Beurteilung mit dem Mikroskop sind, dass die Bilder standortunabhängig beurteilt werden und damit auch leicht Zweitmeinungen eingeholt werden können, dass eine digitale Auswertung und Quantifizierung, z.B. einer immunhistochemischen Färbung, möglich werden und rasch auf archivierte Fälle zugegriffen werden kann.4
Erste kommerzielle Systeme sind verfügbar, allerdings erst in wenigen Instituten etabliert. Längerfristig ist mittels „machine learning“ und „arteficial intelligence“ die Implementierung von Computer-basierten Entscheidungshilfen zu erwarten, beispielsweise für die automatisierte Detektion von Mikrometastasen in Lymphknoten oder das Tumorgrading.
Akkreditierung ist das Maß aller Dinge
Seit Mai 2017 gilt die In-vitro-Diagnostika(IVD)-Verordnung der Europäischen Union und bis Mai 2022 müssen medizinische Laboratorien Anforderungen an die Qualität und Kompetenz nach der ISO-Norm 15189:2012 erfüllen und nach dieser Normvorgabe akkreditiert sein. Diese über eine ISO-Zertifizierung hinausgehenden Anforderungen an die Etablierung eines umfassenden Qualitätsmanagement- und Dokumentationssystems sind derzeit eine große Herausforderung für die Institute für Pathologie in Österreich. Das Ziel der IVD-Verordnung ist die Sicherstellung von europaweit einheitlichen hohen Qualitätsstandards in der Labordiagnostik.
Fazit
Es ist zu erwarten, dass die nächsten 20 Jahre in der Pathologie eine breite Implementierung der digitalen Pathologiebringen werden, zunächst vor allem für die Befundung von Geweben am Computerschirm und die Archivierung der histologischen Bilder. Die genetischen Analysen werden weiter zunehmen und wahrscheinlich werden, bei Reduktion der derzeit für die klinische Diagnostik noch zu hohen Fehlerrate, als „third-generation sequencing“ bezeichnete Technologien noch einmal einen Quantensprung in Bezug auf Einfachheit der Probenbearbeitung, Umfang der sequenzierten Gene und Schnelligkeit der Analysen bringen. Die Zukunft der Pathologie ist morpho-molekular. Zu hoffen ist, dass die Pathologie ihr fachliches Spektrum über die derzeit dominierende Tumorpathologie hinaus wieder verbreitern kann und die große Tradition als „Lehre von den Krankheiten“ fortsetzt.
Literatur:
1 van den Tweel JG, Taylor CR: A brief history of pathology: Preface to a forthcoming series that highlights milestones in the evolution of pathology as a discipline. Virchows Arch 2010; 457(1): 3-10 2 Heather JM, Chain B: The sequence of sequencers: The history of sequencing DNA. Genomics 2016; 107(1): 1-8 3 Wan JCM et al.: Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nature Reviews Cancer 2017; 17(4): 223-38 4 Grobholz R.: Digitale Pathologie. Der Pathologe 2018; 39(3): 228-35
Das könnte Sie auch interessieren:
Highlights zu Lymphomen
Assoc.Prof. Dr. Thomas Melchardt, PhD zu diesjährigen Highlights des ASCO und EHA im Bereich der Lymphome, darunter die Ergebnisse der Studien SHINE und ECHELON-1
ASH 2020 – Highlights zu den aggressiven Lymphomen
Highlight-Themen der virtuellen ASH-Jahrestagung im Dezember 2020 waren an erster Stelle die Immunonkologika in all ihren Variationen, aber auch Beispiele für innovative Sequenztherapien ...
Aktualisierte Ergebnisse für Blinatumomab bei neu diagnostizierten Patienten
Die Ergebnisse der D-ALBA-Studie bestätigen die Chemotherapie-freie Induktions- und Konsolidierungsstrategie bei erwachsenen Patienten mit Ph+ ALL. Mit einer 3-jährigen ...