Klinische Implikationen der Mikrobiomforschung
Autor:
Dr. med. Christoph Stein-Thoeringer
Abteilung Mikrobiom und Krebs
Deutsches Krebsforschungszentrum
Heidelberg
Nationales Centrum für Tumorerkrankungen
Heidelberg
Sie sind bereits registriert?
Loggen Sie sich mit Ihrem Universimed-Benutzerkonto ein:
Sie sind noch nicht registriert?
Registrieren Sie sich jetzt kostenlos auf universimed.com und erhalten Sie Zugang zu allen Artikeln, bewerten Sie Inhalte und speichern Sie interessante Beiträge in Ihrem persönlichen Bereich
zum späteren Lesen. Ihre Registrierung ist für alle Unversimed-Portale gültig. (inkl. allgemeineplus.at & med-Diplom.at)
Das Mikrobiom des Magen-Darm-Traktes ist ein hochkomplexes Ökosystem. Wenn es aus dem Gleichgewicht gerät, betreffen die Auswirkungen nicht nur den Magen-Darm-Trakt, sondern den gesamten Organismus. Von grosser klinischer Relevanz ist dies vor allem im Zusammenhang mit Antibiotikatherapien, da diese eine der wichtigsten Ursachen für Störungen des Mikrobioms sind. Ein verantwortungsvoller Umgang mit der Verschreibung von Antibiotika hilft somit auch, Schäden am Mikrobiom zu verhindern, die ihrerseits Krankheitsprozesse und andere Therapien negativ beeinflussen können.
Zu den Grundlagen des Mikrobioms
Der erwachsene menschliche Körper beherbergt ungefähr 1013 Bakterien, was etwa der Anzahl menschlicher Zellen entspricht.1 Dieses mikrobielle Ökosystem, das allgemein als Mikrobiom bezeichnet wird, setzt sich zusätzlich zu Bakterien auch aus Eukaryoten (Pilze und Protozoen), Viren und Prokaryoten zusammen2 und ist in ein umfassendes Spektrum an physiologischen und pathologischen Prozessen des Menschen involviert. Die 100-fach höhere Gendiversität des Mikrobioms codiert diverse Mechanismen und Stoffwechselkompetenzen, die ihre eigene mikrobielle Nische sowie Wirtshomöostase und Pathologie beeinflussen können.3,4 Die Mehrheit der kommensalen Bakterien besiedelt den Magen-Darm-Trakt (hier primär das Kolon), aber auch andere anatomische Regionen wie Mund, Atemwegs- und Urogenitaltrakt, Haut oder auch Gewebe wie Tumoren werden durch kommensale Bakterien besiedelt. Insgesamt existieren Wirt und Mikrobiom unter physiologischen Bedingungen in einem symbiotischen Gleichgewicht als Metaorganismus, indem der Wirt im Gegenzug für mikrobielle Unterstützung von Verdauungs- und Stoffwechselvorgängen eine nährstoffreiche Umgebung im Gastrointestinaltrakt bereitstellt. Das Mikrobiom als solches synthetisiert zum Beispiel Vitamine und zerlegt die Nahrung in absorbierbare Nährstoffe wie Kohlenhydrate, kurz- und langkettige Fettsäuren oder andere Metabolite, die wiederum dem Mensch für seinen Metabolismus oder die Immunregulation zugutekommen.5
Zahlreiche epidemiologisch angelegte Studien konnten zeigen, dass Unterschiede im geografischen Standort, in der ethnischen und sozioökonomischen Zugehörigkeit und in den Ernährungsgewohnheiten dazu führen, dass das menschliche Mikrobiom zwischen und innerhalb von Individuen sehr variabel ist.6,7 Unter den physiologischen Faktoren zeigte sich zudem in unabhängigen Untersuchungen, dass vor allem Alter, Geschlecht, der Body-Mass-Index (BMI, hier besonders Übergewicht), Nierenfunktion (gemessen mittels glomerulärer Filtrationsrate, GFR), Alkoholkonsum und diverse diätetische Faktoren den stärksten Einfluss auf die Zusammensetzung des intestinalen Mikrobioms bei gesunden erwachsenen Menschen hat.6,8
Mikrobiom und Krankheitsprozesse
In den letzten zwanzig Jahren hat sich die Erforschung des Mikrobioms zu einem Gebiet von enormem Interesse für eine breite wissenschaftliche Gemeinschaft entwickelt, was primär auf der Entwicklung und praktischen Verfügbarkeit von Hochdurchsatz-Gensequenzierungsmethoden («next-generation sequencing») des Mikrobioms, u.a. der 16S-rRNA-Gensequenzierung, und der Implementierung von bioinformatischen Algorithmen zur standardisierten Mikrobiomanalytik beruht. Zudem wurden grosse Kohortenstudien und Konsortien wie das Human Microbiome Project 1/2 (USA), das American Gut Project (USA), MetaHIT (EU) oder das IHMS-Projekt (EU) gegründet, um das Mikrobiom, seine in- und extrinsischen Einflussfaktoren und seine Rolle bei Krankheitsprozessen systematisch und populationsweit zu untersuchen. In all den bisherigen Studien zu Mikrobiomveränderungen und Krankheitsentstehung haben sich zahlreiche Assoziationen mit Herz-Kreislauf-Erkrankungen,9 entzündlichen Darmerkrankungen,10 Diabetes mellitus,11 kardiometabolischen Erkrankungen,12 Lebererkrankungen,13 neurologischen Entwicklungsstörungen14 und insbesondere Krebserkrankungen15 mit Veränderungen des Darmmikrobioms oder der Mikroben-Wirt-Interaktionen, die als Dysbiose bezeichnet werden, gezeigt. Die bislang erforschten Mechanismen der Mikrobiom-Wirt-Interaktionen in der Entstehung von Krankheiten weisen auf eine direkte Rolle von kommensalen Bakterien als potenziell pathogene Mikroben hin, die beispielsweise über sezernierte bakterielle Proteine wie Lipopolysaccharid (LPS) die Entstehung von Autoimmunität16 oder via Colibactin-Genotoxizität17 und somit Karzinogenese im Gastrointestinaltrakt massgeblich beeinflussen können. Andere Mechanismen beruhen auf der Verstoffwechslung von Nahrungsbestandteilen. So begünstigt zum Beispiel der Verzehr von rotem Fleisch und L-Carnitin die Bildung von mikrobiellen Metaboliten als Vorstufen von Trimethylamin-N-Oxid, welches beim Menschen die Bildung von Atherosklerose verstärken kann.18 Darüber hinaus gibt es noch zahlreiche weitere pathophysiologische Beispiele der Mikrobiom-Wirts-Interaktion, wobei das Spektrum noch nicht vollständig erforscht und wissenschaftlich gesichert ist.
Einsatz von Antibiotika als klinisch relevanter Faktor für die Dysbiose des Mikrobioms
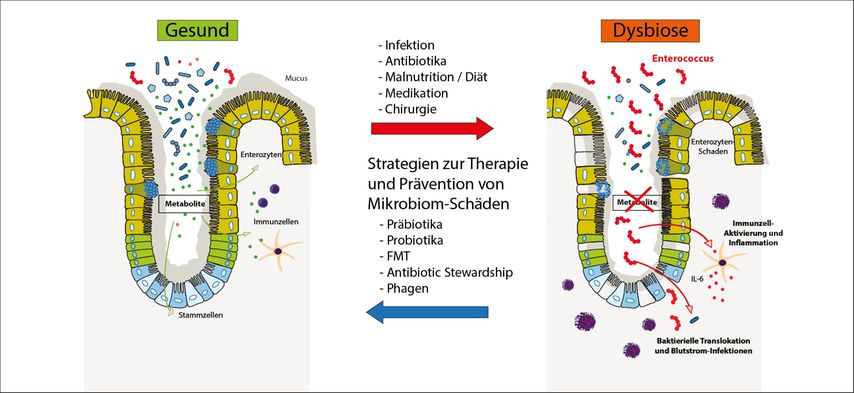
Die Behandlung mit Antibiotika ist eine der Hauptursachen für die Störung des menschlichen Darmmikrobioms. Veränderungen in der mikrobiellen Zusammensetzung sind abhängig von der Dosierung, der Dauer der Behandlung, der Form der Anwendung und der Klasse der Antibiotika. In verschiedenen Studien wurden die kurz- und langfristigen Auswirkungen auf das Darmmikrobiom während und nach einer Antibiotikabehandlung untersucht. Die häufigste Beobachtung war eine Abnahme der Alpha-Diversität des Mikrobioms. So wurde beispielsweise gezeigt, dass kurzzeitige Gaben des Chinolons Ciprofloxacin oder von Meropenem plus Vancomycin bei gesunden Probanden die Diversität des Mikrobioms bis zu 6 Monate lang stören kann.19,20 In einer anderen Studie wurde darüber hinaus festgestellt, dass die Verabreichung von Probiotikamixturen nach Gabe der Antibiotika Ciprofloxacin plus Metronidazol bei gesunden Probanden die Wiederherstellung von Mikrobiomdiversität nicht beschleunigen kann, sondern im Gegenteil sogar weiter verzögert und eine mikrobielle Dysbiosis im Gastrointestinaltrakt verstärken kann.21 Die Ernährung wurde als weiterer Faktor für eine gestörte Erholung nach Antibiotikastress identifiziert und hierbei wurde festgestellt, dass ballaststofffreie Ernährung eine antibiotikainduzierte Dysbiose des Darmmikrobioms protrahiert und ebenso das intestinale Mikromilieu und Metabolom stört.22
Klinisch wurden hierbei epidemiologische Zusammenhänge zwischen Antibiotikatherapien im Kleinkindalter und erhöhtem Risiko für Asthma festgestellt, wobei hierbei signifikante Assoziationen mit veränderter Alpha-Diversität in den ersten Lebensjahren festgestellt wurden.23 In ähnlicher Weise prädisponiert die Antibiotikatherapie im Kleinkindalter für die Entwicklung von Übergewicht im späteren Leben,24 was in präklinischen Experimenten auf eine Dysbiose des intestinalen Mikrobioms und gleichzeitig metabolische Störungen zurückgeführt werden konnte.25 Im Erwachsenenalter führen Antibiotikagaben ebenfalls zu Mikrobiom-assoziierten Kollateralschäden, die von infektiologischen Erkrankungen bis zur Krebsmedizin reichen. Antibiotikatherapien können beispielsweise die Kolonisationsresistenz des intestinalen Mikromilieus reduzieren und Infektionen mit Clostridioides difficile, Klebsiella pneumoniae oder E. coli begünstigen.26,27 Am anderen Ende des Spektrums stehen T-Zell-basierte Immuntherapien gegen solide maligne Erkrankungen. Hier wurde wiederholt beschrieben, dass die Verabreichung von Breitspektrum-Antibiotika bei Krebspatienten das intestinale Mikrobiom schädigt und signifikante Assoziationen mit einer verminderten Effektivität der Immuntherapie sowie einer erhöhten Mortalität gefunden wurden.28 Gleiche Zusammenhänge wurden für die allogene Stammzelltransplantation (allo-HCT) oder CD19-CAR-T-Zelltherapien gegen maligne hämatologische Neoplasien beobachtet.29,30 Pathophysiologisch versucht man sich diese Zusammenhänge so zu erklären, dass durch Antibiotikatherapien eine Reihe von kommensalen Darmbakterien, wie zum Beispiel Bifidobacterium- oder Clostridium-Spezies, eradiziert werden, die mikrobielle Metaboliten wie Adenosin oder kurzkettige Fettsäuren im Darm produzieren, die für die Anti-Tumor-Funktion von humanen T-Zellen entscheidend sind.31,32
Das intestinale Mikrobiom als Effektorstruktur («druggable target») im klinischen Kontext
Die Verabreichung von Antibiotika ist in den allermeisten klinischen Situationen zur Therapie von Infektionen oder auch Prävention von infektiologischen Komplikationen nach Operationen oder Transplantationen (allo-HCT) unvermeidbar. Um den Kollateralschaden auf das Mikrobiom zu begrenzen, werden aktuell verschiedene Strategien untersucht. Wenngleich noch kontrovers diskutiert, gibt es doch ernst zu nehmende Evidenz, dass Probiotikatherapien die Erholung des Mikrobioms nach Antibiotikastress nicht begünstigen und sogar vermindern.21,33 Im Gegensatz dazu führt eine autologe fäkale Mikrobiota-Transplantation (FMT) mit intestinalem Mikrobiommaterial, das vor der Antibiotikatherapie gesammelt, prozessiert und gelagert wurde, zu einer vollständigen und raschen Erholung des patientenspezifischen Mikrobioms nach Antibiotikatherapien.21,34 Weitere alternative Strategien, die aktuell in klinischen Phase-II/III-Studien getestet werden, beruhen darauf, Antibiotika im Darm per se über aktivierte Nanopartikel35 oder Beta-Lactamasen36 zu neutralisieren, sodass bei erhaltener systemischer Antibiotikaeffektivität der Mikrobiomschaden reduziert wird.
Die FMT per se wurde als eine der wichtigsten Interventionsstrategien in der klinisch-translationalen Mikrobiomforschung in zahlreichen Indikationen getestet und zeigt v.a. bei infektiologischen und entzündlichen Erkrankungen des Darms wie der Clostridioides-difficile-Infektion (CDI), der Colitis ulcerosa und der intestinalen Graft-vs.-Host-Disease nach allo-HCT sehr gute und reproduzierbare Wirksamkeit.37–39 Im Speziellen zeigte sich in Phase-I/II-Studie auch, dass sogar die Wirksamkeit von Immuncheckpoint-Immuntherapien durch die FMT von Mikrobiommaterial von Immuntherapie-Respondern erhöht werden kann.40,41 Nicht nur komplexe mikrobielle Konstrukte wie die FMT zeigen eine klinische Wirkung in der Immuntherapie, sondern auch Probiotika wie Bifidobakterien, deren orale Verabreichung die Wirkung von Nivolumab plus Ipilimumab auf das progressionsfreie Überleben von Nierenzellkarzinompatienten signifikant erhöht hat.42 Allgemein gibt es jedoch bis heute Unklarheit, was die Effektorwirkung der FMT betrifft – seien es nun vermehrungsfähige, vitale Mikroben oder ihre Metaboliten, da in Pilotstudien gezeigt wurde, dass sterile FMT-Filtrate, die mikrobielle Stoffwechselprodukte oder Bakteriophagen enthalten, ebenso gegen CDI oder die nekrotisierende Enterokolitis wirksam sind.43,44 Eine Zusammenfassung von klinisch relevanten Interventionsstrategien in der translationalen Mikrobiomforschung zeigt Abbildung 1.
Zusammenfassung
Obgleich wir erst am Anfang der Erforschung des Wechselspiels von Mikrobiom und Mensch stehen, kann man bereits feststellen, dass das Mikrobiom in zahlreiche Erkrankungen involviert ist und auch die Wirksamkeit von klinisch etablierten Therapien von der Homöostase des Mikrobioms abhängig ist. In diesem Zusammenhang ist auf die Verschreibungspraxis betreffend Antibiotika hinzuweisen, da aus klinischer Perspektive nicht nur der Aspekt von «Antibiotic Stewardship» zu beachten ist. Es ist vielmehr aufgrund der zunehmenden klinischen Bedeutung des humanen Mikrobioms auch zu bedenken, dass Antibiotika, insbesondere mit Breitspektrumaktivität, Schäden am Mikrobiom und in der Mikrobe-Wirt-Interaktion induzieren, die Krankheitsprozesse oder klinische Therapien negativ beeinflussen können, sodass in diesem Zusammenhang auch von einem «Microbiome Stewardship» zu sprechen ist.
Abb. 2: Der erwachsene menschliche Körper beherbergt ungefähr 1013 Bakterien, was etwa der Anzahl menschlicher Zellen entspricht
Literatur:
1 Sender R et al.: PLoS Biol 2016; 14: e1002533 2 Kern L et al.: Curr Opin Microbiol 2021; 63: 158-71 3 Ansaldo E et al.: Annu Rev Immunol 2021; 39: 449-79 4 Koh A, Backhed F: Mol Cell 2020; 78: 584-96 5 Lin W et al.: Curr Opin Microbiol 2021; 63: 150-7 6 Vujkovic-Cvijin I et al.: Nature 2020; 587: 448-54 7 Gacesa R et al.: Nature 2022; 604: 732-9 8 Byrd, AL et al.: J Exp Med 2021; 218: e20200606 9 Fromentin S et al.: Nat Med 2022; 28: 303-14 10 Lloyd-Price J et al.: Nature 2019; 569: 655-62 11 Vatanen T et al.: Nature 2018; 562: 589-94 12 Koeth RA et al.:J Clin Invest 2019; 129: 373-87 13 Wang R et al.: Cell Mol Immunol 2021; 18: 4-17 14 Morais LH et al.: Nat Rev Microbiol 2021; 19: 241-55 15 Cullin N et al.:Cancer Cell 2021; 39: 1317-41 16 Vatanen T et al.: Cell 2016; 165: 842-53 17 Pleguezuelos-Manzano C et al.: Nature 2020; 580: 269-73 18 Koeth RA et al.: Nat Med 2013; 19: 576-85 19 Dethlefsen L, Relman DA: Proc Natl Acad Sci U S A 2011; 108 Suppl 1: 4554-61 20 Palleja A et al.: Nat Microbiol 2018; 3: 1255-65 21 Suez J et al.: Cell 2018; 174: 1406-23 22 Tanes C et al.: Cell Host Microbe 2021; 29: 394-407 23 Patrick DM et al.: Lancet Respir Med 2020; 8: 1094-105 24 Leong KSW et al.: JAMA Netw Open 2020; 3: e1919681 25 Mahana D et al.: Genome Med 2016; 8: 48 26 Sorbara MT et al.: J Exp Med 2019; 216: 84-98 27 Buffie CG et al.: Nature 2015; 517: 205-08 28 Pinato DJ et al.: JAMA Oncol 2019; 5: 1774-8 29 Shono Y et al.: Sci Transl Med 2016; 8: 339ra371 30 Jenq RR et al.: Biol Blood Marrow Transplant 2015; 21: 1373-83 31 Mager LF et al.: Science 2020; 369: 1481-9 32 Docampo MD et al.: Blood 2022; 139: 2392-405 33 Montassier E et al.: Nat Microbiol 2021; 6: 1043-54 34 Taur Y et al.: RSci Transl Med 2018; 10: eaap9489 35 de Gunzburg J et al.: J Infect Dis 2018; 217: 628-36 36 Kokai-Kun JF et al.: Lancet Infect Dis 2019; 19: 487-96 37 van Nood E et al.: N Engl J Med 2013; 368: 407-15 38 Costello SP et al.: JAMA 2019; 321: 156-64 39 Goeser F et al.: Eur J Haematol 2021; 107: 229-45 40 Baruch EN et al.: Science 2021; 371: 602-9 41 Davar D et al.: Science 2021; 371: 595-602 42 Dizman N et al.: Nat Med 2022; 28: 704-12 43 Ott SJ et al.: Gastroenterology 2017; 152: 799-811 44 Brunse A et al.: ISME J 2022; 16: 686-94
Das könnte Sie auch interessieren:
Neues aus der Gastroenterologie
Nicht jede Alkoholisierung ist auf Alkoholkonsum zurückzuführen. Beim sogenannten Eigenbrauer-Syndrom kommt es infolge pathologischer Auffälligkeiten des Darmmikrobioms zur endogenen ...
Zöliakie: Stand der Entwicklungpharmakologischer Therapieoptionen
Viele Patienten mit Zöliakie haben trotz glutenfreier Diät weiterhin Beschwerden. Zurzeit befinden sich einige innovative therapeutische Ansätze in Entwicklung: u.a. ein Inhibitor der ...
Das kardiovaskuläre Risiko von IBD-Patienten
Eine aktive IBD erhöht das Risiko für kardiovaskuläre Erkrankungen, während bestehende kardiovaskuläre Probleme die Wahl der Medikation erschweren. Das Ziel ist es, die richtige Balance ...